前两天有小伙伴在问免疫浸润分析,半夜睡不着起来码字写一下免疫细胞浸润分析。聊聊为什么要做免疫浸润分析,并介绍如何使用R包 CIBERSORT 进行免疫浸润分析,以及如何解读分析结果。
为什么要做免疫浸润分析?
肿瘤微环境(Tumor Microenvironment, TME)是肿瘤细胞与其周围免疫细胞、基质细胞等共同构成的复杂生态系统。免疫细胞在肿瘤微环境有很多作用:一方面,它们可以识别并清除肿瘤细胞;另一方面,肿瘤细胞也可以通过多种机制逃避免疫系统的监视,甚至“驯化”免疫细胞为其生长和转移提供支持。
免疫细胞浸润分析的核心目标是量化肿瘤组织中各类免疫细胞的相对比例。通过这种分析,我们可以:
-
评估肿瘤的免疫状态 :了解肿瘤微环境中免疫细胞的组成,判断是否存在免疫抑制或免疫激活状态。 -
预测患者预后 :某些免疫细胞(如细胞毒性T细胞)的高浸润通常与较好的预后相关,而调节性T细胞(Tregs)或肿瘤相关巨噬细胞(TAMs)的高浸润可能与较差的预后相关。 -
指导免疫治疗 :免疫检查点抑制剂(如PD-1/PD-L1抑制剂)的疗效与肿瘤微环境中的免疫细胞浸润密切相关。通过免疫浸润分析,可以筛选出更适合免疫治疗的患者。
CIBERSORT做免疫浸润分析的数据原理
CIBERSORT 是一种基于基因表达数据的反卷积算法,能够从复杂的组织表达谱中推断出22种免疫细胞亚型的相对比例。它的核心思想是通过已知的免疫细胞特征矩阵,将混合表达数据分解为不同免疫细胞的贡献。
CIBERSORT的核心是线性最小二乘回归(Linear Least Squares Regression)。其数学模型可以表示为:
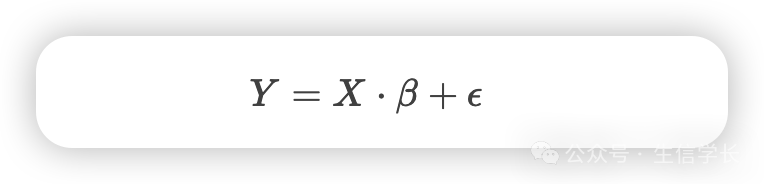
其中:
-
Y 是观察到的组织基因表达矩阵(混合信号) -
X 是已知的免疫细胞特征矩阵(每列代表一种免疫细胞的基因表达特征) -
β 是待求解的免疫细胞比例矩阵 -
ϵ 是误差项
CIBERSORT通过优化算法求解 β ,使得 Y 与 X 之间的差异最小化,从而得到每种免疫细胞在样本中的相对比例。
为了确保结果的可靠性,CIBERSORT还通过置换检验,重复多次引入 P值 来评估反卷积结果的显著性。P值越低,说明反卷积结果越可信。
如何使用R包CIBERSORT进行免疫浸润分析?
接下来,我们使用R包 CIBERSORT 对基因表达矩阵进行免疫浸润分析。
1. 安装和加载CIBERSORT
首先,确保你已经安装了R和必要的依赖包。然后,下载CIBERSORT的R脚本(可从官网获取)并加载。
# 引用所需的R包
library("limma") # 加载limma包用于基因表达数据处理
# 读取输入文件并对数据进行整理
data <- read.table("expression_matrix.txt", header = TRUE, sep = "\t", check.names = FALSE, row.names = 1) # 读取表达数据文件
data <- data[rowMeans(data) > 0,] # 过滤掉平均表达量为0的基因
# 打印表达矩阵
data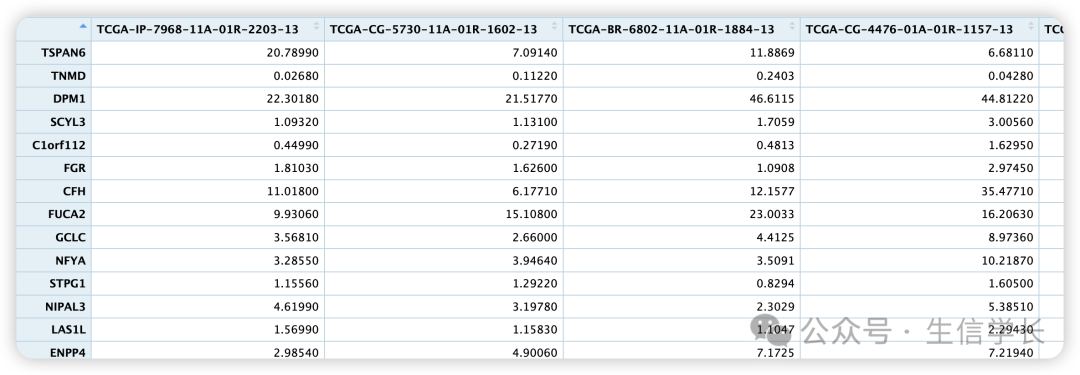
2. 运行CIBERSORT分析
使用
CIBERSORT
函数对表达矩阵进行反卷积分析。
# 加载CIBERSORT脚本
source("CIBERSORT.R")
# 运行CIBERSORT
results <- CIBERSORT(lm22_matrix, expr_matrix, perm=100, QN=TRUE)
# 查看结果
results
-
perm:设置置换次数,用于计算P值; -
QN:是否对数据进行分位数标准化。
免疫浸润的结果如下
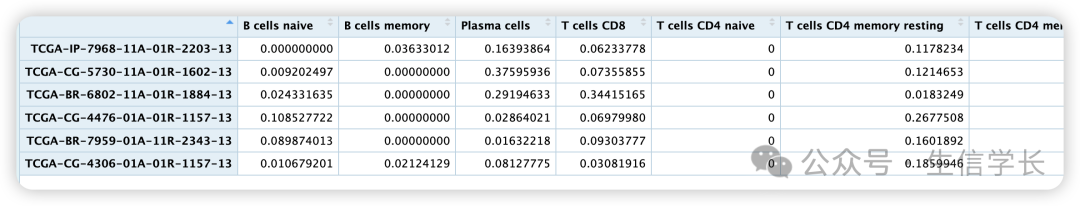
1. 免疫细胞比例
-
B细胞 、 T细胞 、 巨噬细胞 等免疫细胞的比例反映了肿瘤微环境的免疫状态。 -
例如,高比例的细胞毒性T细胞(CD8+ T cells) 通常与较好的预后相关,而高比例的 调节性T细胞(Tregs)可能与免疫抑制相关。
2. P值
-
P值小于0.05通常被认为是显著的,表明反卷积结果可信。
代码&文件联系老师获取

写作不易,欢迎关注
