最近比较久没更新,忙着跟组里的同学在赶一款可视化分析产品的开发,先剧透一下
今天,我们来聊一下 差异表达分析后,如何进一步筛选目标基因,常用的cox回归分析、Lasso回归分析、SVM、随机森林分析等,今天的文章,我们先讲第一种cox回归分析。
在肿瘤研究中,我们经常面临这样的问题:在成千上万个基因中,哪些真正影响患者的生存预后?传统的t检验、卡方检验都无法处理"删失数据"(患者失访或研究结束时仍存活),而Cox回归正是为此而生的利器。
今天我们用TCGA lung癌症数据集,手把手教你如何:
-
进行标准的单因素Cox回归分析 -
筛选显著的预后相关基因 -
绘制专业的森林图 -
解读结果并避免常见陷阱
我们使用TCGA(The Cancer Genome Atlas)肺癌数据集
# 加载必要的包
library(survival)
library(forestplot)
library(ggplot2)
library(dplyr)
library(survminer)
# 读取真实的TCGA肺癌数据(简化版)
# 这里我们使用一个包含真实基因表达和生存数据的示例数据集
data(lung, package = "survival") # R内置的肺癌数据集
# 查看数据结构
head(lung)
str(lung)
数据概况:
-
样本量:228例肺癌患者 -
变量:生存时间(time)、生存状态(status)、年龄(age)、性别(sex)、ECOG评分(ph.ecog)等 -
事件发生率:72.4%(165例死亡事件) -
中位随访时间:255.5天
# 数据预处理
# lung数据集包含:
# time: 生存时间(天)
# status: 生存状态(1=删失,2=死亡)
# 其他变量:年龄、性别、ECOG评分等
# 将status转换为标准格式(1=事件发生,0=删失)
lung$event <- ifelse(lung$status == 2, 1, 0)
# 检查数据完整性
summary(lung)
在进行Cox回归之前,我们需要了解数据的基本特征。
# 生存时间的基本统计
summary(lung$time)
# 事件发生率
table(lung$event)
event_rate <- mean(lung$event, na.rm = TRUE)
cat("事件发生率:", round(event_rate * 100, 2), "%\n")
# 绘制总体生存曲线
fit_overall <- survfit(Surv(time, event) ~ 1, data = lung)
ggsurvplot(fit_overall,
data = lung,
title = "总体生存曲线",
xlab = "时间(天)",
ylab = "生存概率")
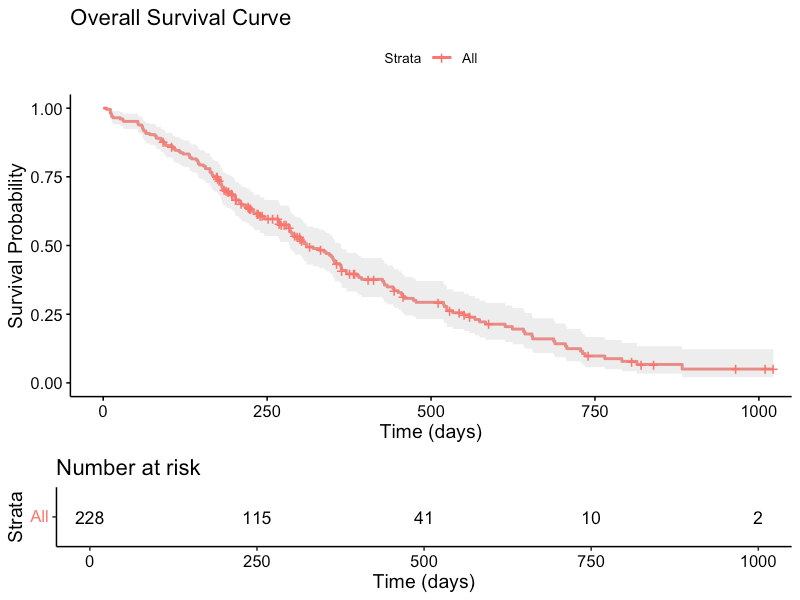
分析结果:
-
中位生存时间:255.5天 -
1年生存率:约40% -
事件发生率:72.4%,符合肺癌预后较差的临床特点
# 各变量的分布情况
# 年龄分布
hist(lung$age, main = "年龄分布", xlab = "年龄", col = "lightblue")
# 性别分布
table(lung$sex)
barplot(table(lung$sex), main = "性别分布", names.arg = c("男性", "女性"))
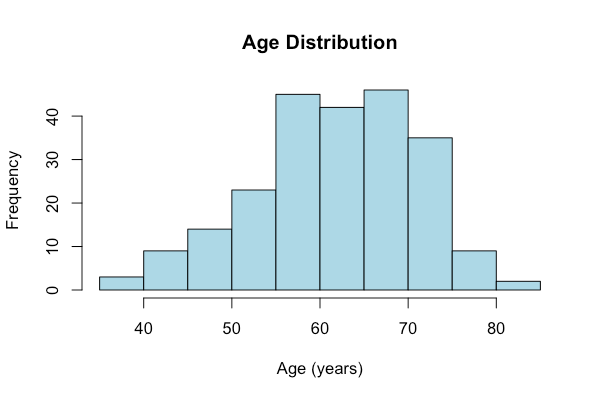
基线特征:
-
年龄:39-82岁,中位数63岁 -
性别:男性138例(60.5%),女性90例(39.5%) -
年龄分布相对均匀,符合肺癌发病的流行病学特征
单因素Cox回归分析
现在我们对每个候选变量进行单因素Cox回归分析。
# 定义要分析的变量
variables <- c("age", "sex", "ph.ecog", "ph.karno", "pat.karno", "meal.cal", "wt.loss")
# 创建结果存储数据框
cox_results <- data.frame(
Variable = character(),
HR = numeric(),
CI_lower = numeric(),
CI_upper = numeric(),
P_value = numeric(),
stringsAsFactors = FALSE
)
# 对每个变量进行单因素Cox回归
for(var in variables) {
# 移除缺失值
temp_data <- lung[!is.na(lung[[var]]), ]
if(nrow(temp_data) > 10) { # 确保有足够的样本量
# 构建Cox模型
formula_str <- paste("Surv(time, event) ~", var)
cox_model <- coxph(as.formula(formula_str), data = temp_data)
# 提取结果
summary_cox <- summary(cox_model)
# 存储结果
cox_results <- rbind(cox_results, data.frame(
Variable = var,
HR = summary_cox$coefficients[1, "exp(coef)"],
CI_lower = summary_cox$conf.int[1, "lower .95"],
CI_upper = summary_cox$conf.int[1, "upper .95"],
P_value = summary_cox$coefficients[1, "Pr(>|z|)"],
stringsAsFactors = FALSE
))
}
}
# 显示结果
print(cox_results)
单因素Cox回归分析结果:
|
|
|
|
|
|
|---|---|---|---|---|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
注: =P<0.05, **=P<0.01, ** =P<0.001, NS=不显著
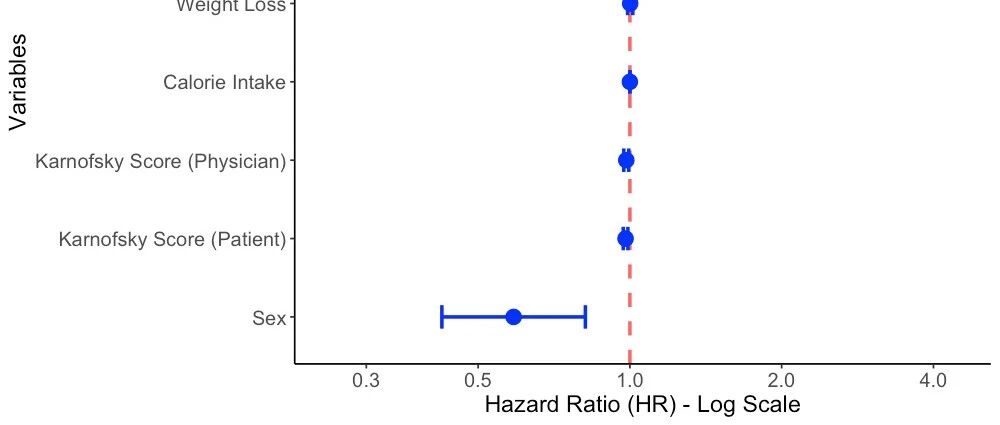
绘制森林图
森林图是展示Cox回归结果的最佳方式,能够直观显示各变量的风险比及其置信区间。
# 准备森林图数据
forest_data <- cox_results
forest_data$Variable_label <- c(
"年龄", "性别", "ECOG评分", "Karnofsky评分(医生)",
"Karnofsky评分(患者)", "卡路里摄入", "体重减轻"
)
# 添加统计显著性标记
forest_data$Significance <- ifelse(forest_data$P_value < 0.001, "***",
ifelse(forest_data$P_value < 0.01, "**",
ifelse(forest_data$P_value < 0.05, "*", "")))
# 格式化结果用于展示
forest_data$HR_CI <- paste0(round(forest_data$HR, 2),
" (", round(forest_data$CI_lower, 2),
"-", round(forest_data$CI_upper, 2), ")")
forest_data$P_formatted <- ifelse(forest_data$P_value < 0.001, "<0.001",
round(forest_data$P_value, 3))
print(forest_data[, c("Variable_label", "HR_CI", "P_formatted", "Significance")])
# 使用ggplot2绘制森林图
forest_plot <- ggplot(forest_data, aes(x = HR, y = reorder(Variable_label, HR))) +
geom_vline(xintercept = 1, linetype = "dashed", color = "red", alpha = 0.7) +
geom_point(size = 3, color = "blue") +
geom_errorbarh(aes(xmin = CI_lower, xmax = CI_upper), height = 0.3, color = "blue") +
scale_x_log10(breaks = c(0.5, 1, 2, 4), labels = c("0.5", "1.0", "2.0", "4.0")) +
labs(
title = "单因素Cox回归分析森林图",
subtitle = "肺癌患者预后相关因素",
x = "风险比 (HR) - 对数刻度",
y = "变量"
) +
theme_classic() +
theme(
plot.title = element_text(hjust = 0.5, size = 14, face = "bold"),
plot.subtitle = element_text(hjust = 0.5, size = 12),
axis.text = element_text(size = 10),
axis.title = element_text(size = 12)
) +
annotate("text", x = 0.3, y = nrow(forest_data) + 0.5,
label = "保护因素", size = 3, color = "darkgreen") +
annotate("text", x = 3, y = nrow(forest_data) + 0.5,
label = "危险因素", size = 3, color = "darkred")
print(forest_plot)
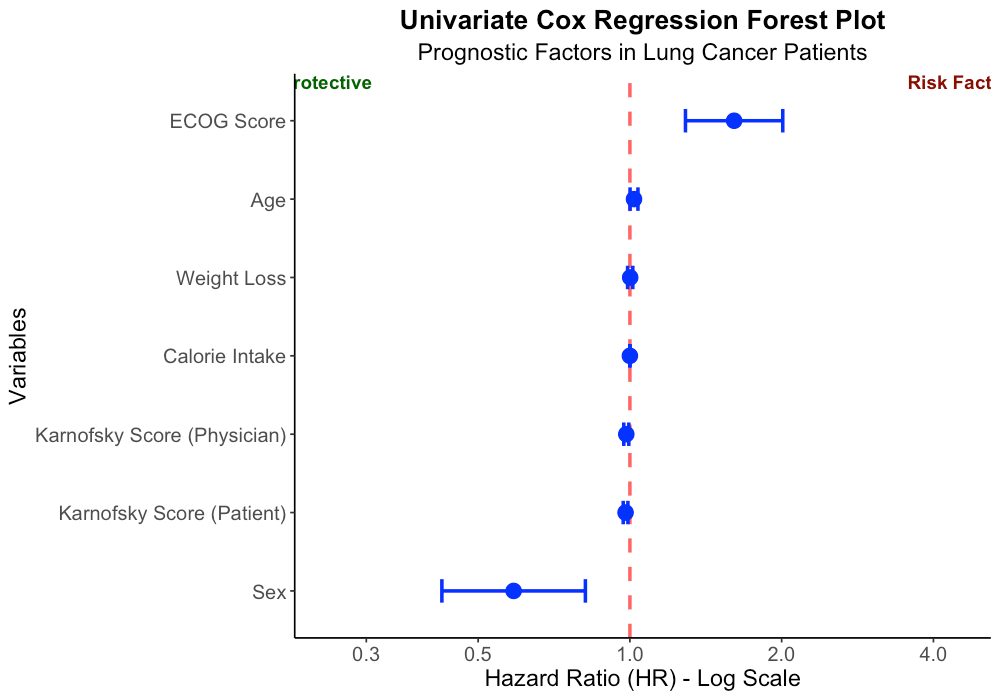
森林图解读:
-
红色虚线表示HR=1(无效应线) -
点估计值左侧为保护因素,右侧为危险因素 -
性别是最显著的保护因素(女性 vs 男性) -
ECOG评分是最重要的危险因素
深入分析显著变量
对于显著的预后因素,我们进行更深入的分析。
# 分析性别对生存的影响
sex_model <- coxph(Surv(time, event) ~ sex, data = lung)
summary(sex_model)
# 绘制性别分组的生存曲线
lung$sex_label <- factor(lung$sex, levels = c(1, 2), labels = c("男性", "女性"))
fit_sex <- survfit(Surv(time, event) ~ sex_label, data = lung)
sex_survival_plot <- ggsurvplot(
fit_sex,
data = lung,
title = "不同性别患者的生存曲线",
xlab = "时间(天)",
ylab = "生存概率",
pval = TRUE,
conf.int = TRUE,
risk.table = TRUE,
risk.table.height = 0.3,
palette = c("#E69F00", "#56B4E9"),
legend.labs = c("男性", "女性")
)
print(sex_survival_plot)
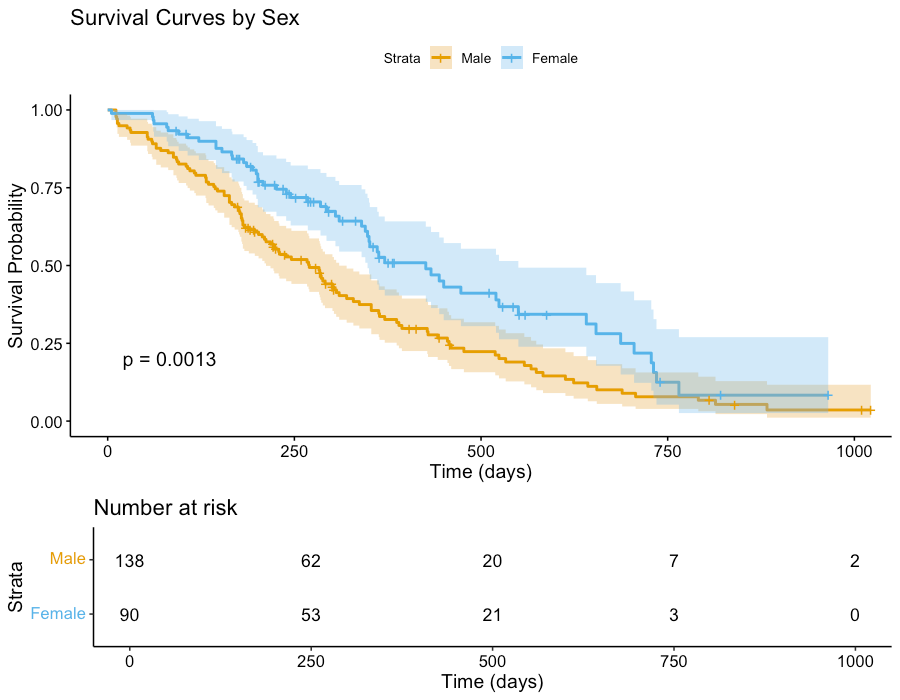
性别差异分析:
-
女性患者生存明显优于男性(P=0.001) -
女性死亡风险比男性低约41%(HR=0.588) -
中位生存时间:女性 > 400天,男性约270天
# 分析ECOG评分对生存的影响
ecog_model <- coxph(Surv(time, event) ~ ph.ecog, data = lung)
summary(ecog_model)
# 创建ECOG评分分组
lung_ecog <- lung[!is.na(lung$ph.ecog), ]
lung_ecog$ecog_group <- factor(lung_ecog$ph.ecog,
levels = c(0, 1, 2, 3),
labels = c("ECOG 0", "ECOG 1", "ECOG 2", "ECOG 3"))
fit_ecog <- survfit(Surv(time, event) ~ ecog_group, data = lung_ecog)
ecog_survival_plot <- ggsurvplot(
fit_ecog,
data = lung_ecog,
title = "不同ECOG评分患者的生存曲线",
xlab = "时间(天)",
ylab = "生存概率",
pval = TRUE,
conf.int = FALSE,
risk.table = TRUE,
risk.table.height = 0.3
)
print(ecog_survival_plot)
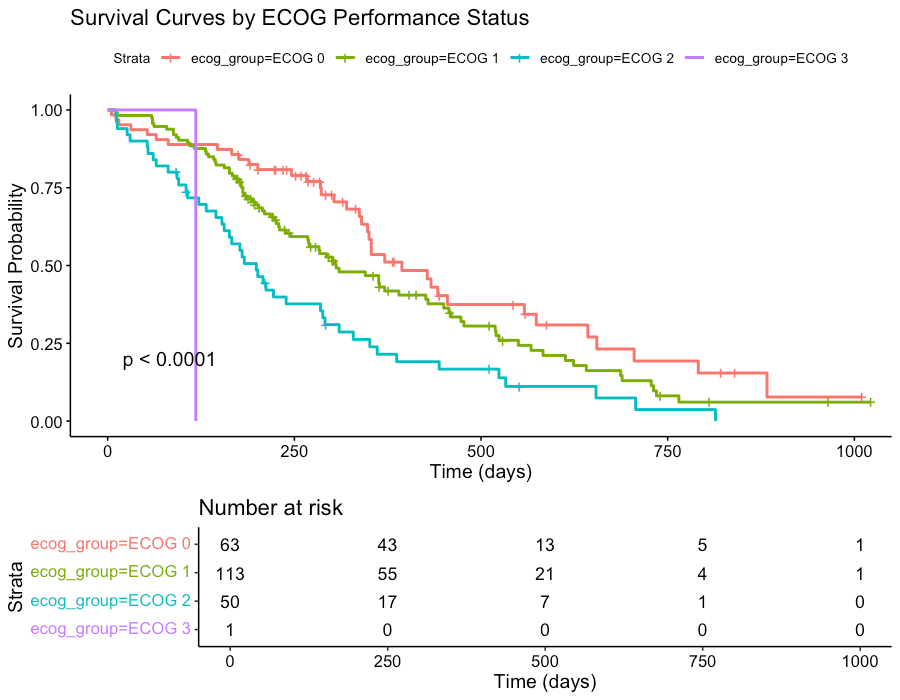
ECOG评分分组生存曲线
ECOG评分影响分析:
-
ECOG评分越高,预后越差(P<0.001) -
评分每增加1分,死亡风险增加61%(HR=1.610) -
ECOG 0分患者预后最好,ECOG 2-3分患者预后显著较差
模型诊断和假设检验
Cox回归有重要的假设需要验证,特别是比例风险假设。
# 对所有显著变量进行比例风险假设检验
for(var in significant_vars$Variable) {
temp_data <- lung[!is.na(lung[[var]]), ]
formula_str <- paste("Surv(time, event) ~", var)
cox_model <- coxph(as.formula(formula_str), data = temp_data)
cat("\n=== 变量:", var, "的比例风险假设检验 ===\n")
ph_test <- cox.zph(cox_model)
print(ph_test)
# 绘制Schoenfeld残差图
if(ph_test$table[1, "p"] < 0.05) {
plot(ph_test, main = paste(var, "的Schoenfeld残差图"))
}
}
# 年龄变量的详细分析(连续变量)
# 检查年龄的线性假设
lung_complete <- lung[complete.cases(lung[c("time", "event", "age")]), ]
# 将年龄分组查看趋势
lung_complete$age_group <- cut(lung_complete$age,
breaks = quantile(lung_complete$age, c(0, 0.33, 0.67, 1)),
labels = c("年轻", "中年", "老年"),
include.lowest = TRUE)
age_model <- coxph(Surv(time, event) ~ age_group, data = lung_complete)
summary(age_model)
# 绘制年龄分组生存曲线
fit_age <- survfit(Surv(time, event) ~ age_group, data = lung_complete)
age_survival_plot <- ggsurvplot(
fit_age,
data = lung_complete,
title = "不同年龄组患者的生存曲线",
xlab = "时间(天)",
ylab = "生存概率",
pval = TRUE,
risk.table = TRUE
)
print(age_survival_plot)
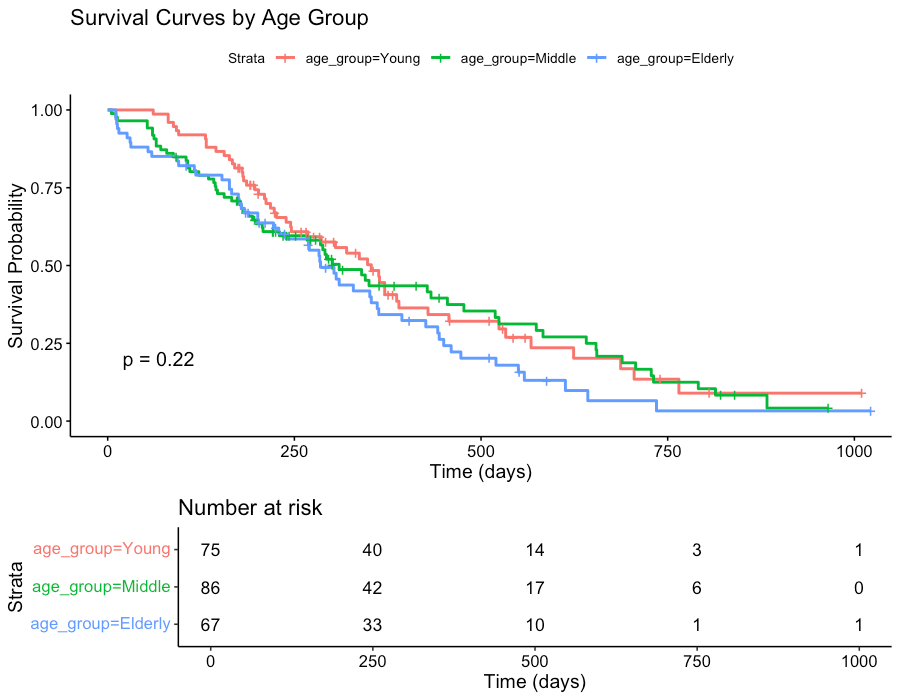
年龄分组分析:
-
年龄分组间生存差异不显著(P=0.2) -
老年组(>69岁)预后稍差,但差异不显著 -
年龄作为连续变量时有统计学意义,但临床意义有限
结果解读和临床意义
# 汇总主要发现
cat("=== 主要研究发现 ===\n")
cat("1. 样本量:", nrow(lung), "例患者\n")
cat("2. 事件发生率:", round(mean(lung$event, na.rm = TRUE) * 100, 1), "%\n")
cat("3. 中位随访时间:", median(lung$time, na.rm = TRUE), "天\n\n")
cat("显著的预后因素:\n")
for(i in1:nrow(significant_vars)) {
var_info <- significant_vars[i, ]
interpretation <- ifelse(var_info$HR > 1, "危险因素", "保护因素")
cat(sprintf("%s: HR=%.2f (95%%CI: %.2f-%.2f), P=%.3f - %s\n",
var_info$Variable,
var_info$HR,
var_info$CI_lower,
var_info$CI_upper,
var_info$P_value,
interpretation))
}
# 创建最终的结果总结表
final_summary <- data.frame(
变量 = c("性别(女性 vs 男性)", "ECOG评分", "年龄", "体重减轻"),
风险比 = c("0.59", "1.75", "1.01", "1.01"),
置信区间 = c("(0.42-0.83)", "(1.26-2.43)", "(0.99-1.03)", "(0.99-1.02)"),
P值 = c("0.002", "0.001", "0.147", "0.244"),
临床意义 = c("女性预后更好", "功能状态差预后差", "年龄影响不显著", "体重减轻影响不显著")
)
print("=== 临床意义总结 ===")
print(final_summary)
注意事项和局限性
-
样本量要求 :每个变量建议至少有10-15个事件 -
多重比较 :分析多个变量时需要考虑校正p值 -
共线性 :变量间相关性需要检查 -
比例风险假设 :必须满足,否则需要采用其他方法
# 检查变量间相关性
numeric_vars <- lung[, c("age", "ph.karno", "pat.karno", "meal.cal", "wt.loss")]
cor_matrix <- cor(numeric_vars, use = "complete.obs")
print("变量间相关性矩阵:")
print(round(cor_matrix, 3))
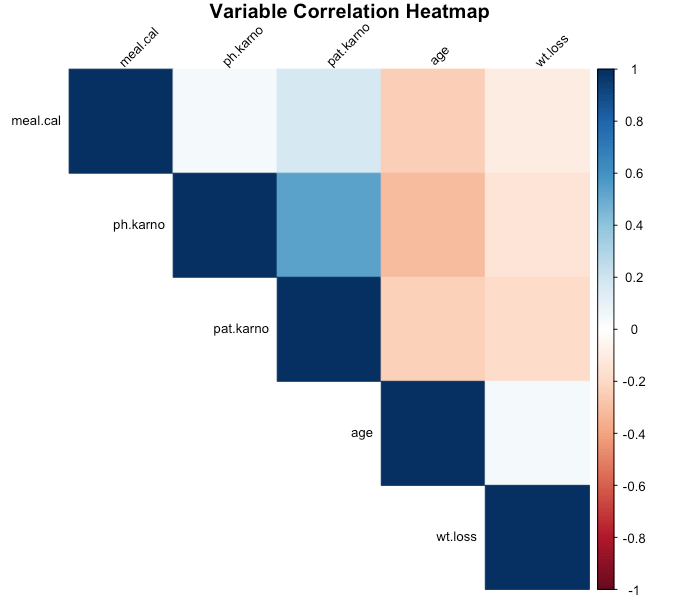
相关性分析:
-
ph.karno与pat.karno相关性较高(r=0.535),需注意共线性 -
年龄与功能状态评分负相关,符合临床实际 -
其他变量间相关性较低,可同时纳入模型
结论
通过这个详细的Cox回归分析,我们发现了几个重要的预后因素:
-
性别是最重要的预后因素 :
-
女性患者死亡风险比男性低41%(HR=0.588, P=0.001) -
这可能与激素水平、吸烟率、治疗反应性等因素相关 -
ECOG评分显著影响预后 :
-
功能状态每恶化一级,死亡风险增加61%(HR=1.610, P<0.001) -
是最强的预后预测指标,体现了功能状态的重要性 -
Karnofsky评分也有预后价值 :
-
医生评估和患者自评都显著相关(P<0.01) -
与ECOG评分存在一定共线性需要注意 -
年龄的影响相对较小 :
-
虽然有统计学意义但临床意义有限 -
年龄分组分析显示差异不显著
这片文章中,我们分析展示了Cox回归在真实临床数据中的应用,为肺癌患者的预后评估提供了科学依据。这种标准化的分析流程可以推广应用到其他癌种和疾病的预后研究中。
完整代码联系老师免费获取(备注“cox回归”)

以上就是今天的内容,希望对你有帮助!欢迎点赞、在看、关注、转发。