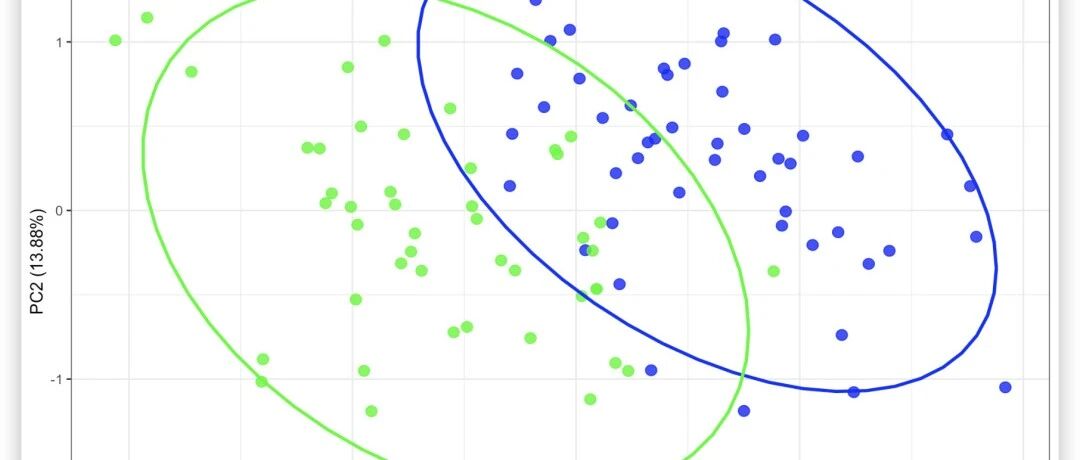
PCA图在生信分析中的应用场景
PCA(主成分分析)是RNA-seq数据分析中最重要的可视化工具之一,主要应用于:
-
样本质量监控 :快速识别实验中的离群样本 -
批次效应检测 :判断数据是否需要批次校正 -
组间差异评估 :观察不同处理组的分离趋势 -
降维可视化 :将上万维度的基因表达数据投影到二维平面 -
聚类结果验证 :辅助判断单细胞数据分群质量
如何解读PCA图
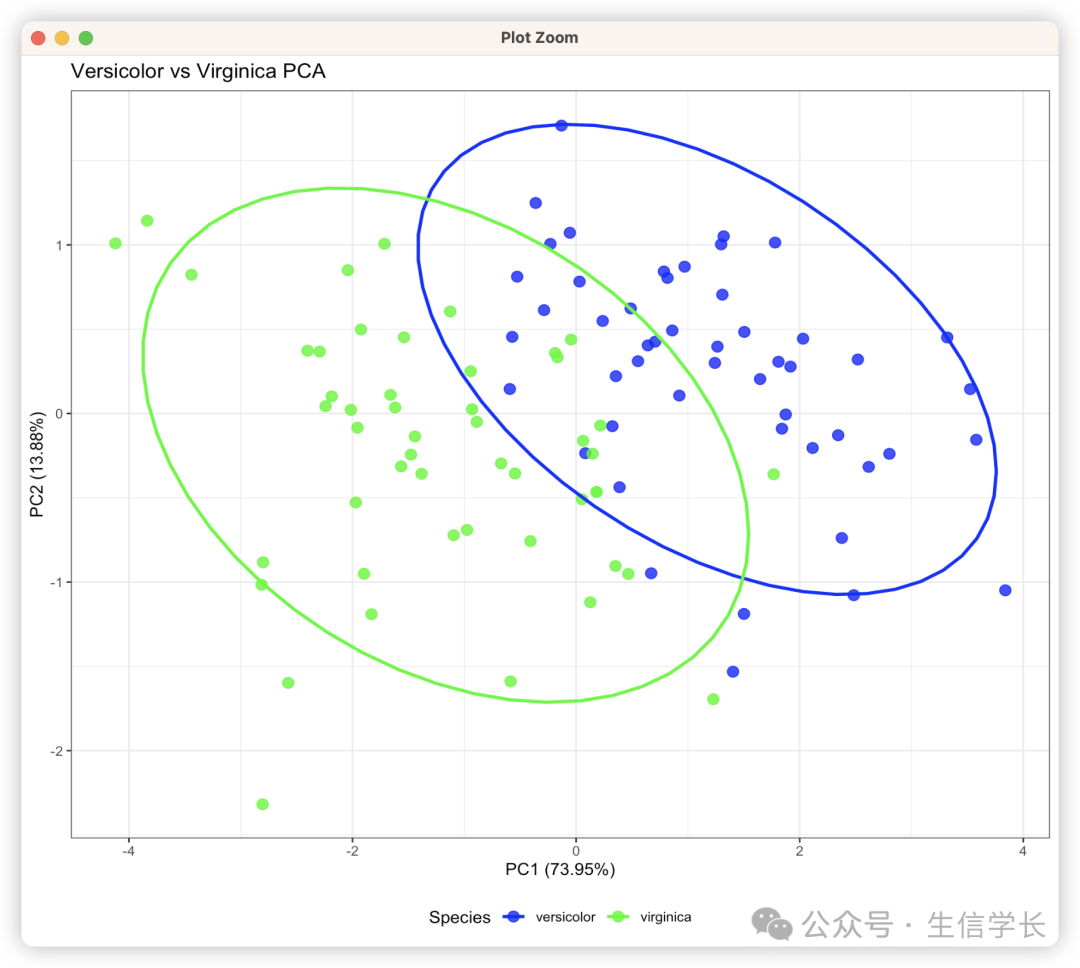
标注说明:
-
坐标轴含义 :X轴(PC1)和Y轴(PC2)代表前两个主成分,括号内百分比表示该成分解释的方差量 -
样本分布 :相同颜色的点代表生物学重复,形状可表示不同实验批次 -
组间距离 :Versicolor和Virginica组在PC1方向的分离差异 -
椭圆区域 :95%置信区间帮助判断 组间差异的显著性
R语言实现教程
data(iris)
# 筛选出versicolor和virginica两类
target_species <- c("versicolor", "virginica")
iris_subset <- subset(iris, Species %in% target_species)
标准化处理
scaled_data <- scale(iris_subset[, 1:4], center = TRUE, scale = TRUE)主成分计算
pca_result <- prcomp(scaled_data, scale. = FALSE)
summary(pca_result)$importance[, 1:2]绘制带置信椭圆的PCA图
library(ggplot2)
library(ggfortify)
# 准备绘图数据
pca_scores <- as.data.frame(pca_result$x)
pca_scores$Species <- iris_subset$Species
# 计算主成分方差百分比
percentage <- round(pca_result$sdev^2 / sum(pca_result$sdev^2) * 100, 2)
x_label <- paste0("PC1 (", percentage[1], "%)")
y_label <- paste0("PC2 (", percentage[2], "%)")
# 绘制带百分比标签的PCA图
ggplot(pca_scores, aes(x = PC1, y = PC2, color = Species)) +
geom_point(size = 3, alpha = 0.8) +
stat_ellipse(level = 0.95, linewidth = 1) +
scale_color_manual(values = c("blue", "green")) +
labs(
title = "Versicolor vs Virginica PCA",
x = x_label, # 添加PC1百分比
y = y_label # 添加PC2百分比
) +
theme_bw() +
theme(legend.position = "bottom")
-
结果解读 :前两个主成分累计解释87%的方差,说明数据具有较好的可解释性 -
生物学启示 :两类花种呈现明显分离趋势,PC1是主要差异来源 -
批次影响 :不同形状的样本点没有明显聚集,提示批次效应较小
完整代码联系老师免费获取(备注“PCA绘图”)

以上就是今天的内容,希望对你有帮助!欢迎点赞、在看、关注、转发。