在高维数据可视化领域,t-SNE曾是降维分析的"黄金标准"。然而,随着数据规模的增长和计算需求的提升,"PCA+UMAP"的两步降维策略正逐渐成为主流选择。这种转变背后有什么技术考量?
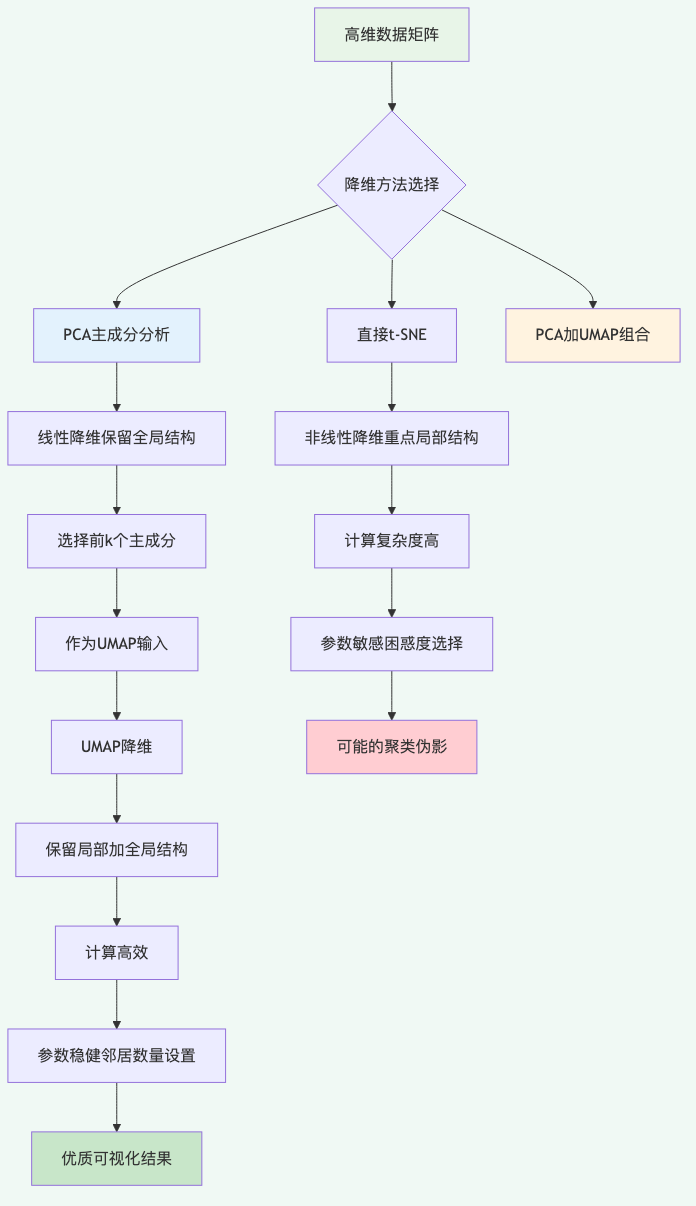
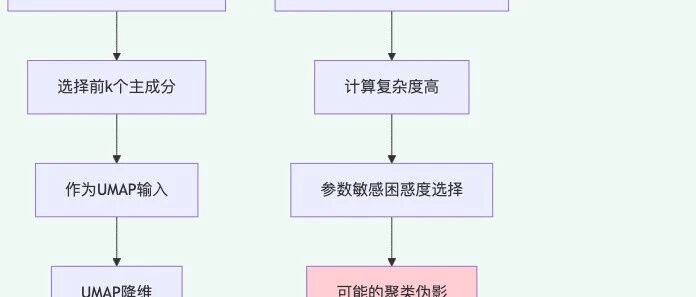
图 1 不同降维方法的技术路线:PCA+UMAP组合展现出更好的综合性能。
计算复杂度的现实挑战
t-SNE的最大限制在于其 O(n²)的计算复杂度 。对于包含数万个细胞的单细胞RNA-seq数据,直接使用t-SNE会面临严重的计算瓶颈。当样本量超过10,000时,内存需求和计算时间都会急剧增长。
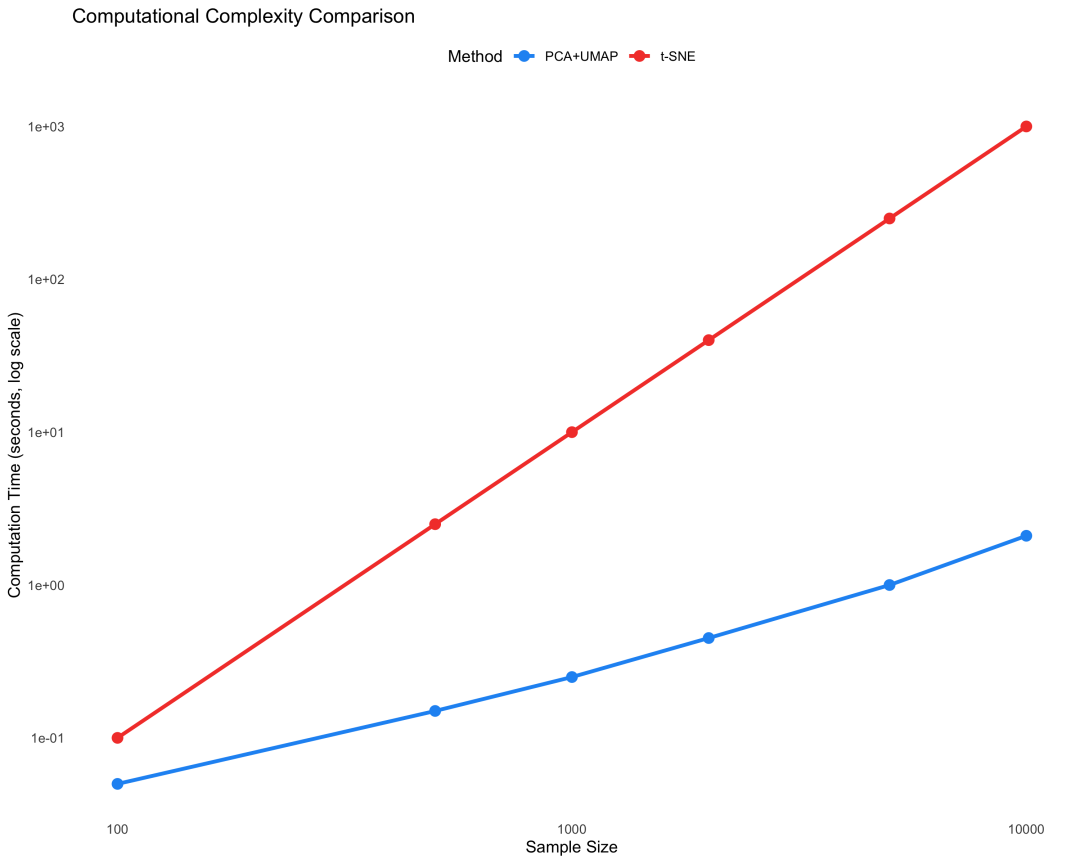
图 2 不同方法的计算时间比较:PCA+UMAP在大规模数据上展现出明显优势。
PCA的线性变换复杂度仅为O(np²),其中p是特征数量。即使对于数万样本,PCA也能在分钟级别完成。UMAP基于近似最近邻算法,复杂度约为O(n log n),在降维后的低维空间中运行更加高效。
全局与局部结构的平衡
PCA保留全局结构 :作为线性方法,PCA能够保持数据的整体拓扑关系,确保样本间的相对距离在降维后仍然有意义。这种全局视野对于理解数据的总体分布模式至关重要。
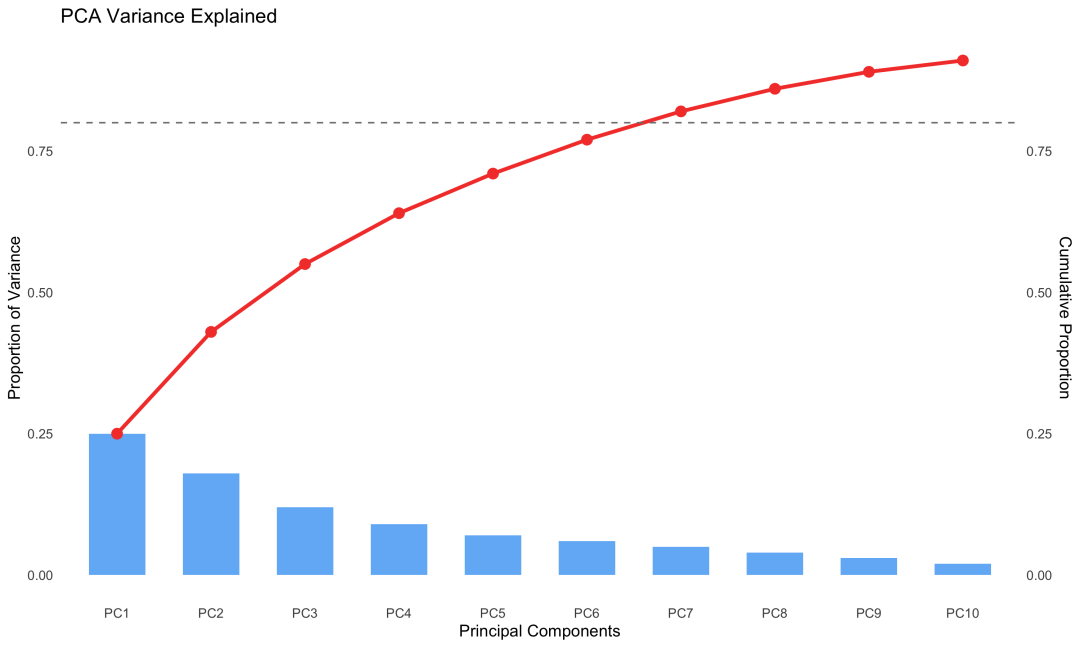
图 3 PCA主成分解释的方差比例:前几个主成分通常能捕获数据的主要变异。
UMAP兼顾两者 :在PCA降维的基础上,UMAP运用流形学习原理,既保留局部邻域结构,又维护一定的全局关系。这种设计使得UMAP能够在聚类清晰度和结构完整性之间找到平衡。
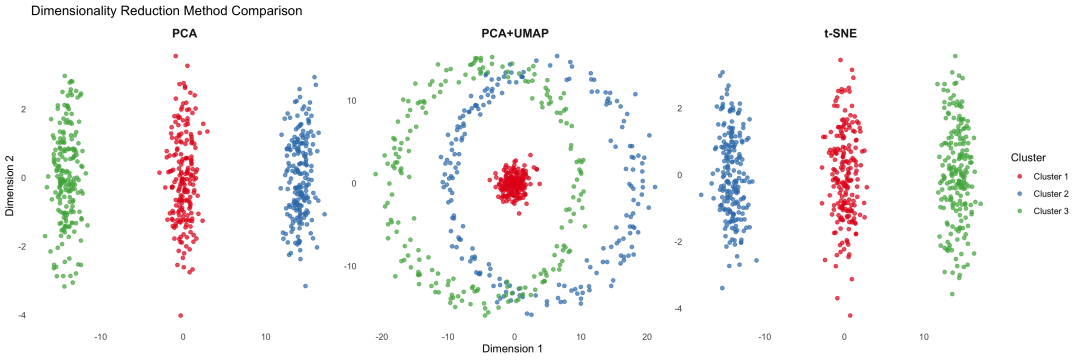
图 4 三种降维方法的可视化效果比较:PCA+UMAP结合了线性和非线性方法的优势。
参数稳定性的实用优势
t-SNE对**困惑度(perplexity)**参数极其敏感,不同的困惑度设置可能产生截然不同的聚类模式。这种敏感性在探索性分析中可能导致误导性结论。
相比之下,UMAP的核心参数(如邻居数量n_neighbors)对结果的影响更加平滑和可预测。PCA更是几乎无需调参,主成分数量的选择主要基于累积方差解释比例的经验阈值(如80%)。
现代组学分析的标准流程
在单细胞基因组学分析中,"PCA+UMAP"已成为标准流程:
-
PCA预处理 :将原始基因表达矩阵降维至50-100个主成分 -
方差过滤 :基于累积贡献率选择有效主成分 -
UMAP可视化 :在主成分空间中进行最终的二维映射 -
聚类分析 :在PCA空间中进行聚类,在UMAP空间中展示
这种流程既保证了计算效率,又维护了生物学解释的可靠性。
结语
PCA+UMAP的组合策略体现了"分而治之"的智慧:用线性方法处理维度爆炸,用非线性方法优化可视化效果。在大数据时代,这种务实的技术选择正推动着生物信息学分析向更高效、更可靠的方向发展。
以上就是今天的内容,希望对你有帮助!欢迎点赞、在看、关注、转发。
写作不易,欢迎关注