今天分享一期有机器学习随机森林来筛选肿瘤,主要展示如何利用R语言构建机器学习模型,基于基因表达数据进行癌症分类,完整代码联系老师免费获取。
一、数据准备与预处理
1.1 加载乳腺癌数据集
我们使用
mlbench
包里面的乳腺癌数据集
# install.packages("caret")
# install.packages("mlbench")
# install.packages("pROC")
# install.packages("ggplot2")
# install.packages("randomForest")
# 加载必要包
library(caret)
library(mlbench)
library(pROC)
library(ggplot2)
library(randomForest)
# 载入乳腺癌数据集
data(BreastCancer)
dim(BreastCancer) # 699个样本 x 11个特征
# 数据预处理
df <- BreastCancer[,-1] # 移除ID列
df$Class <- factor(make.names(ifelse(df$Class == "malignant", 1, 0)))
df <- df[complete.cases(df), ] # 移除缺失值
cat("原始数据维度:", dim(BreastCancer))
cat("处理后数据维度:", dim(df))
运行结果:
原始数据维度:699 x 11
处理后数据维度:683 x 10
1.2 数据标准化与分割
# 划分训练集/测试集
set.seed(123)
trainIndex <- createDataPartition(df$Class, p=0.8, list=FALSE)
train <- df[trainIndex, ]
test <- df[-trainIndex, ]
# 标准化处理(中心化+缩放)
preProc <- preProcess(train[,-10], method=c("center", "scale"))
train_scaled <- predict(preProc, train)
test_scaled <- predict(preProc, test)
可以看到标准化后的训练集和测试集
train_scaled
和
test_scaled
的数据如下

接下来开始跑随机森林机器学习
二、随机森林模型构建
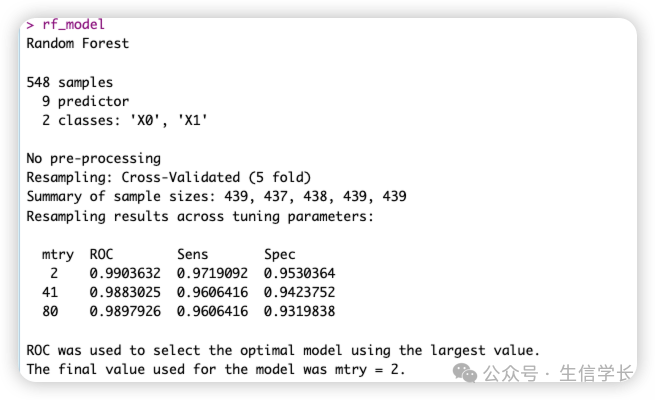
2.1 模型训练
# 设置交叉验证
ctrl <- trainControl(method="cv",
number=5,
classProbs=TRUE,
summaryFunction=twoClassSummary)
# 训练随机森林模型
rf_model <- train(Class ~ .,
data=train_scaled,
method="rf",
trControl=ctrl,
metric="ROC")
上面代码是设置了交叉验证的参数,包括使用5折交叉验证、计算类别概率,并使用
twoClassSummary
函数来评估模型性能,再使用随机森林算法训练模型,并使用前面设置的交叉验证参数来评估模型性能,主要关注ROC曲线下的面积(AUC)。
模型输出:
2.2 模型评估
训练模型后,我们用测试集来验证模型的训练效果
# 测试集预测
pred <- predict(rf_model, newdata=test_scaled)
prob <- predict(rf_model, newdata=test_scaled, type="prob")[,2]
# 混淆矩阵
confusionMatrix(pred, test_scaled$Class)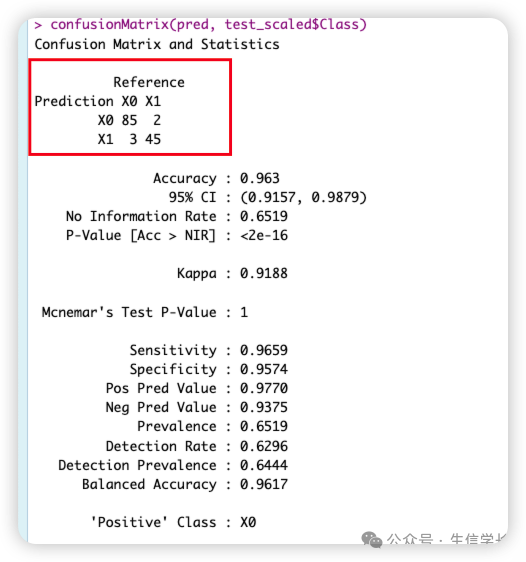
我们可以看到混淆矩阵(红框部分)
-
行(Prediction) :模型的预测结果。 -
列(Reference) :真实的类别标签。 -
X0 和 X1 是两个类别。 -
具体数值 : -
85 :模型正确预测为 X0的样本数(True Positives, TP)。 -
2 :模型错误地将 X1预测为X0的样本数(False Positives, FP)。 -
3 :模型错误地将 X0预测为X1的样本数(False Negatives, FN)。 -
45 :模型正确预测为 X1的样本数(True Negatives, TN)。
以及关键的统计指标
-
Accuracy(准确率) : -
值: 0.963(96.3%)。 -
解释:模型在测试集上的整体预测正确率。 -
95% Confidence Interval(95% 置信区间) : -
值: (0.9157, 0.9879)。 -
解释:准确率的置信区间,表示我们有 95% 的置信度认为真实准确率在这个范围内。 -
No Information Rate(无信息率) : -
值: 0.6519(65.19%)。 -
解释:如果模型总是预测多数类( X0),其准确率会达到 65.19%。这里模型的准确率(96.3%)显著高于无信息率,说明模型性能很好。 -
P-Value [Acc > NIR] : -
值: <2e-16。 -
解释:这是一个假设检验的 p 值,用于检验模型的准确率是否显著高于无信息率。 <2e-16表示模型准确率显著高于无信息率。 -
Kappa(卡帕系数) : -
值: 0.9188。 -
解释:卡帕系数用于衡量模型预测结果与随机预测的一致性。取值范围为 [-1, 1],值越接近 1 表示模型性能越好。0.9188表示模型性能非常好。
最后我们绘制来可视化上面的结果
三、可视化分析
3.1 ROC曲线
# ROC曲线
roc_obj <- roc(test_scaled$Class, prob)
ggroc(roc_obj, color="#4E79A7") +
geom_abline(slope=1, intercept=1, linetype="dashed") +
ggtitle("ROC") +
theme_minimal()
ROC 提用于评估模型的分类性能,我们主要关注 ROC 曲线的形状和 AUC 值,值越接近 1 表示模型性能越好。
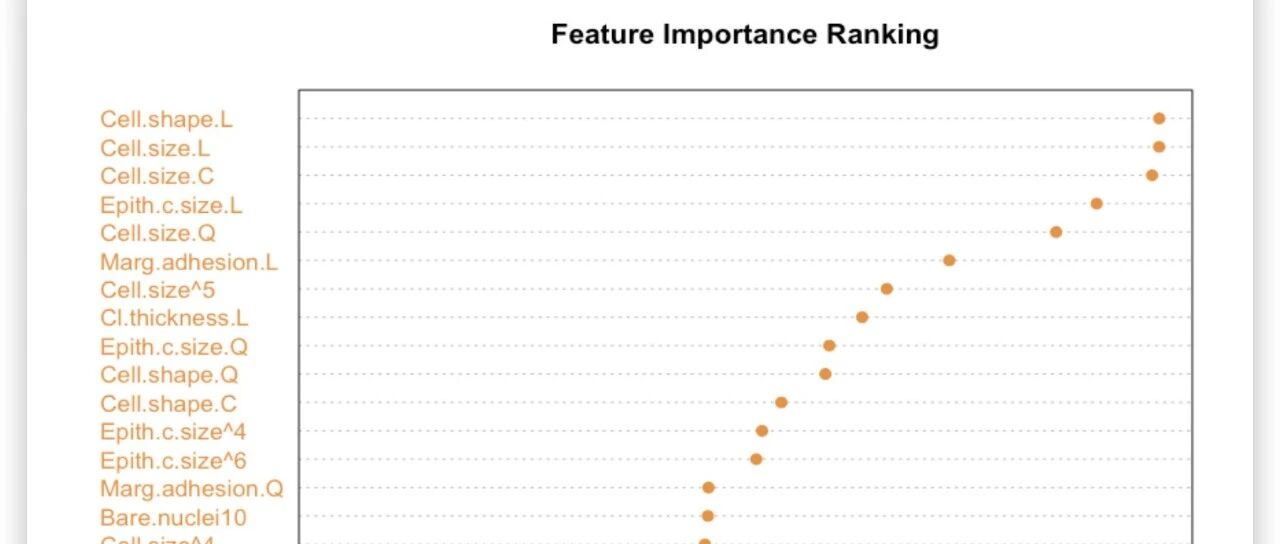
3.2 特征重要性
# 特征重要性
varImpPlot(rf_model$finalModel,
main="Feature Importance Ranking",
col="#F28E2B",
pch=19)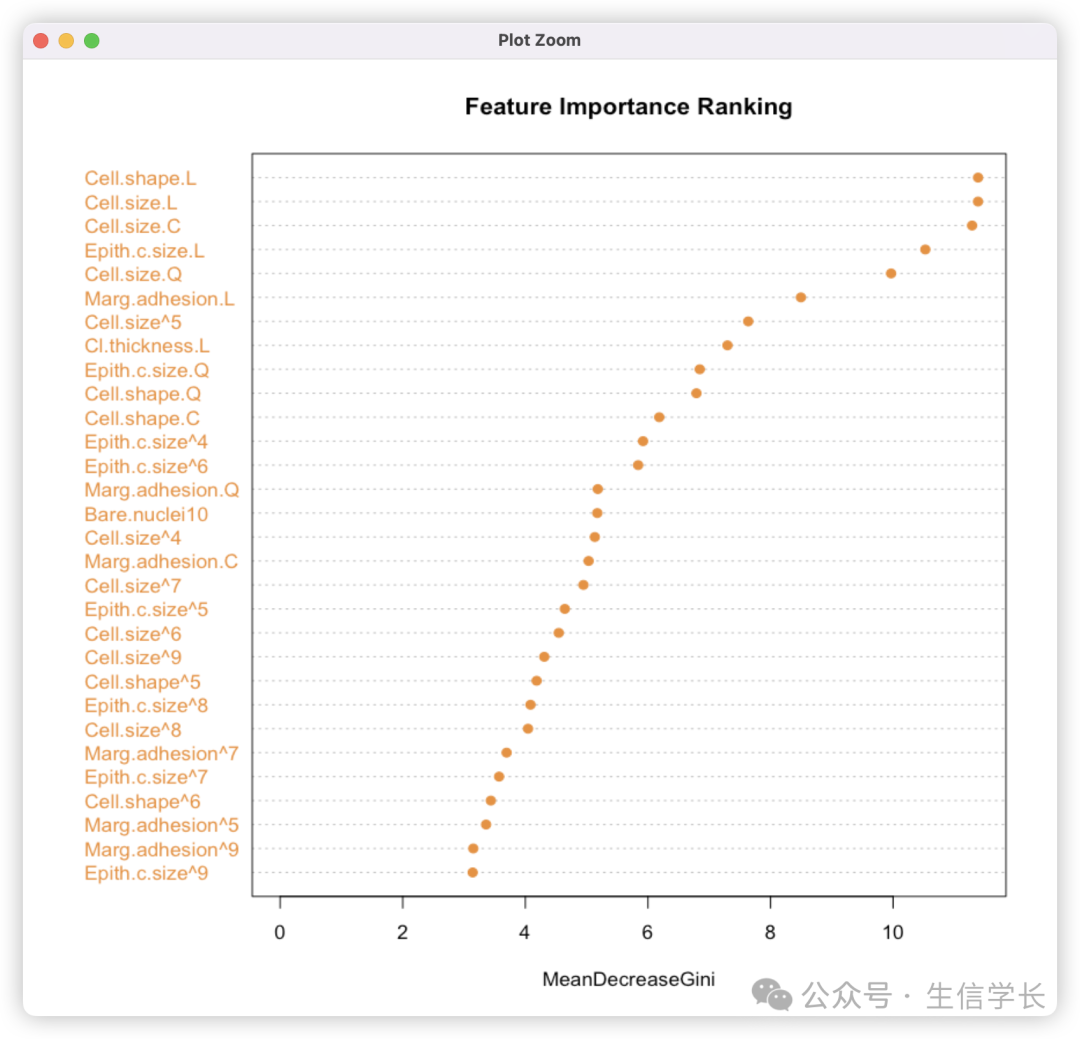
varImp 用于分析随机森林模型中各个特征的重要性。我们主要关注哪些特征的柱子较高,这些特征对模型的预测贡献较大。
总结
我们通过使用乳腺癌数据集,构建随机森林模型来筛选肿瘤样本,主要流程包括完成缺失值处理与标准化,再分割训练集和测试集,使用训练集训练模型,再使用测试集验证,以及最后的ROC,varImp可视化。
完整代码联系老师免费获取(备注“随机森林”)

以上就是今天的内容,希望对你有帮助!欢迎点赞、在看、关注、转发。