点击卡片关注,一起学习生信分析!
什么是ROC曲线?
ROC曲线最初用于雷达信号检测,现在广泛应用于:
-
「医学诊断」 : 评估诊断试验的准确性 -
「机器学习」 : 评估分类模型的性能 -
「预后预测」 : 评估生存预测模型的判别能力
ROC曲线的应用
在生存分析中,ROC曲线可以:
-
评估风险评分的判别能力 -
比较不同预测模型的性能 -
确定最佳截断值(Cut-off) -
评估不同时间点的预测准确性
2. ROC曲线原理
2.1 基本概念
「混淆矩阵」 :
|
|
|
|
|---|---|---|
| 「预测阳性」 |
|
|
| 「预测阴性」 |
|
|
「核心指标」 :
-
「灵敏度(Sensitivity)」 = TP/(TP+FN)
-
正确识别阳性的比例 -
也称真阳性率(TPR) -
「特异度(Specificity)」 = TN/(TN+FP)
-
正确识别阴性的比例 -
「假阳性率(FPR)」 = 1 - 特异度 = FP/(TN+FP)
2.2 ROC曲线绘制
ROC曲线是以**假阳性率(1-特异度)**为X轴,**真阳性率(灵敏度)**为Y轴的曲线。
绘制方法:
-
选择不同的截断值 -
计算每个截断值下的灵敏度和特异度 -
以(1-特异度, 灵敏度)为坐标绘点 -
连接所有点形成ROC曲线
2.3 AUC指标
「AUC」 (Area Under Curve,曲线下面积):
-
范围: 0.5-1.0 -
「0.5」 : 随机猜测,无判别能力 -
「0.5-0.7」 : 较低准确性 -
「0.7-0.9」 : 一定准确性 -
「>0.9」 : 较高准确性
「解释」 : AUC表示随机选择一个阳性样本和一个阴性样本,模型对阳性样本打分高于阴性样本的概率。
2.4 时间依赖ROC
在生存分析中,需要考虑 「时间因素」 :
-
不同时间点的预测准确性可能不同 -
需要计算1年、3年、5年等时间点的ROC -
使用timeROC包实现
3. 环境准备
3.1 R包安装
# 安装必要的R包
install.packages("survival")
install.packages("survminer")
install.packages("timeROC")
install.packages("pROC")
「包的功能」 :
-
pROC: 基本ROC曲线分析 -
timeROC: 时间依赖ROC曲线 -
survival: 生存分析 -
survminer: 生存分析可视化
3.2 数据准备
需要的数据文件:
-
risk.txt: 风险评分数据(来自文章15) -
clinical.txt: 临床信息数据
4. 代码实现
4.1 读取和准备数据
library(pROC)
library(ggplot2)
library(survival)
library(survminer)
library(timeROC)
# 读取风险数据
risk <- read.table("risk.txt",
header = TRUE,
sep = "\t",
check.names = FALSE,
row.names = 1)
# 提取关键列
risk <- risk[, c("time", "state", "riskScore")]
# 读取临床数据
cli <- read.table("clinical.txt",
header = TRUE,
sep = "\t",
check.names = FALSE,
row.names = 1)
# 合并数据
samSample <- intersect(row.names(risk), row.names(cli))
risk1 <- risk[samSample, , drop = FALSE]
cli <- cli[samSample, , drop = FALSE]
rt <- cbind(risk1, cli)
4.2 基本ROC曲线
首先绘制riskScore的基本ROC曲线(二分类):
# 定义颜色
bioCol <- c("DarkOrchid", "Orange2", "MediumSeaGreen", "NavyBlue",
"#8B668B", "#FF4500", "#135612", "#561214")
# ROC分析(生存 vs 死亡)
roc1 <- roc(rt$state ~ rt$riskScore)
# 查看AUC
cat("AUC:", auc(roc1), "\n")
cat("95%CI:", ci.auc(roc1), "\n")
# 创建picture目录
if (!dir.exists("picture")) {
dir.create("picture")
}
# 绘制ROC曲线
png(file = "picture/ROC.riskscore.png",
width = 5 * 300,
height = 5 * 300,
res = 300)
plot(roc1,
print.auc = TRUE,
col = bioCol[1],
legacy.axes = TRUE,
main = "ROC Curve for Risk Score",
lwd = 3)
dev.off()
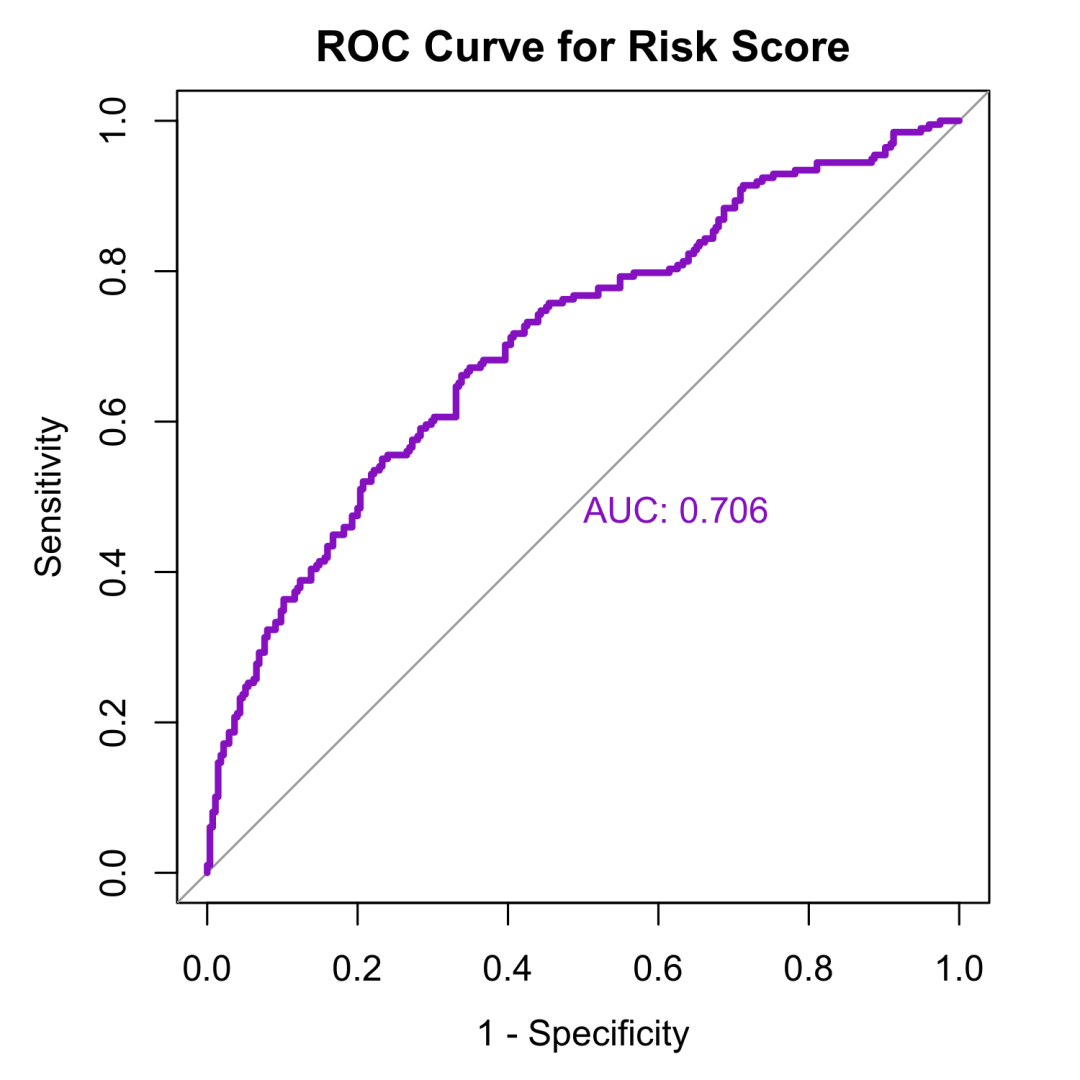
「图形解读」 :
-
「X轴」 : 1 - 特异度(假阳性率) -
「Y轴」 : 灵敏度(真阳性率) -
「紫色曲线」 : riskScore的ROC曲线 -
「对角虚线」 : 随机猜测线(AUC=0.5) -
「AUC = 0.706」 : 表示模型有一定的判别能力
「结果解释」 :
-
AUC = 0.706,说明riskScore对患者预后有较好的判别能力 -
曲线越靠近左上角,判别能力越强 -
95%CI: 0.659-0.754,置信区间不包含0.5,统计学显著
4.3 时间依赖ROC曲线
生存分析中,需要评估不同时间点的预测准确性:
# 计算1年、3年、5年的ROC
ROC_rt <- timeROC(
T = risk$time,
delta = risk$state,
marker = risk$riskScore,
cause = 1,
weighting = 'aalen',
times = c(1, 3, 5),
ROC = TRUE
)
# 查看各时间点的AUC
cat("1年AUC:", ROC_rt$AUC[1], "\n")
cat("3年AUC:", ROC_rt$AUC[2], "\n")
cat("5年AUC:", ROC_rt$AUC[3], "\n")
# 绘制时间依赖ROC曲线
png(file = "picture/ROC.all.png",
width = 5 * 300,
height = 5 * 300,
res = 300)
plot(ROC_rt,
time = 1,
col = bioCol[1],
title = FALSE,
lwd = 4)
plot(ROC_rt,
time = 3,
col = bioCol[2],
add = TRUE,
title = FALSE,
lwd = 4)
plot(ROC_rt,
time = 5,
col = bioCol[3],
add = TRUE,
title = FALSE,
lwd = 4)
legend('bottomright',
c(paste0('AUC at 1 years: ', sprintf("%.03f", ROC_rt$AUC[1])),
paste0('AUC at 3 years: ', sprintf("%.03f", ROC_rt$AUC[2])),
paste0('AUC at 5 years: ', sprintf("%.03f", ROC_rt$AUC[3]))),
col = bioCol[1:3],
lwd = 4,
bty = 'n',
title = "Time-dependent ROC")
dev.off()
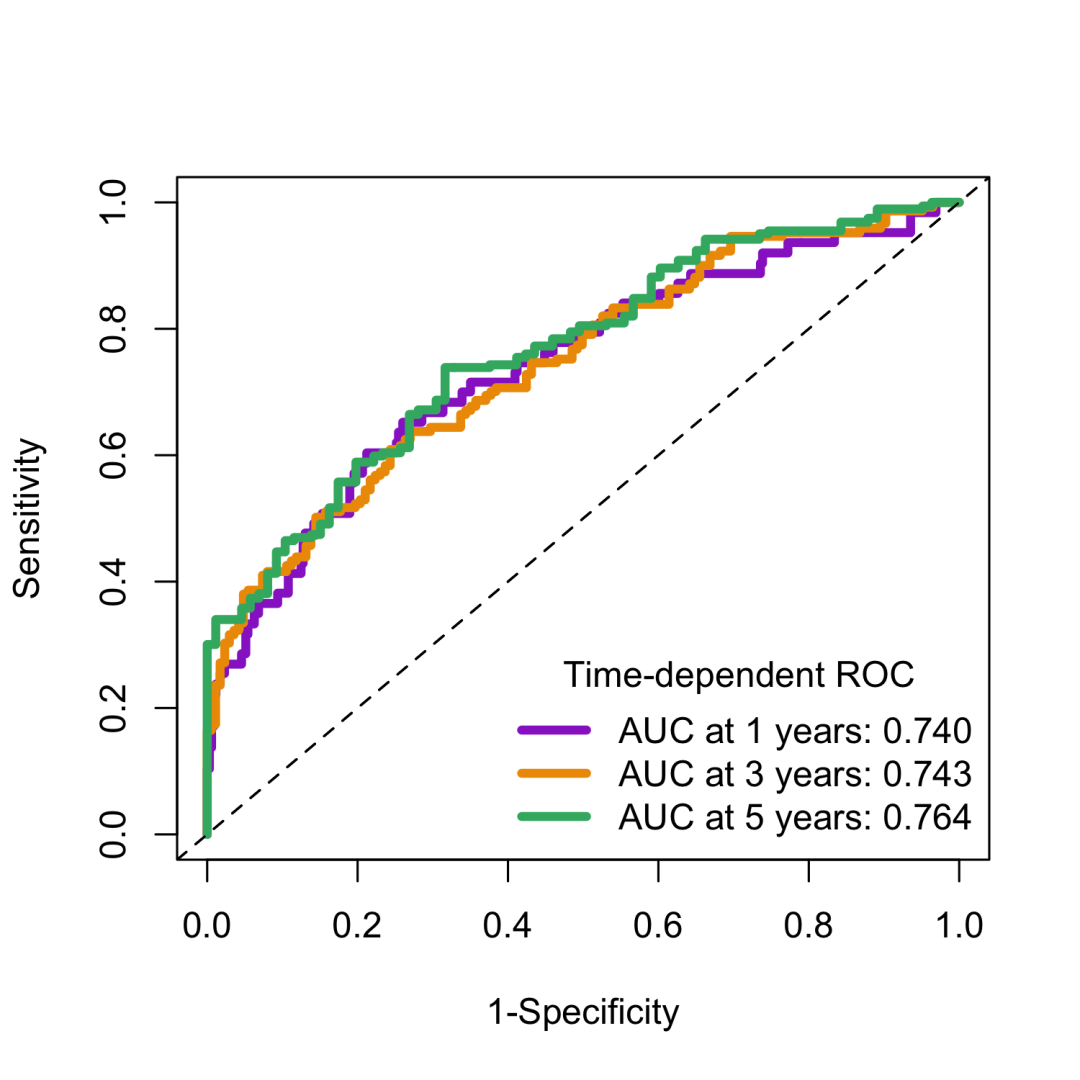
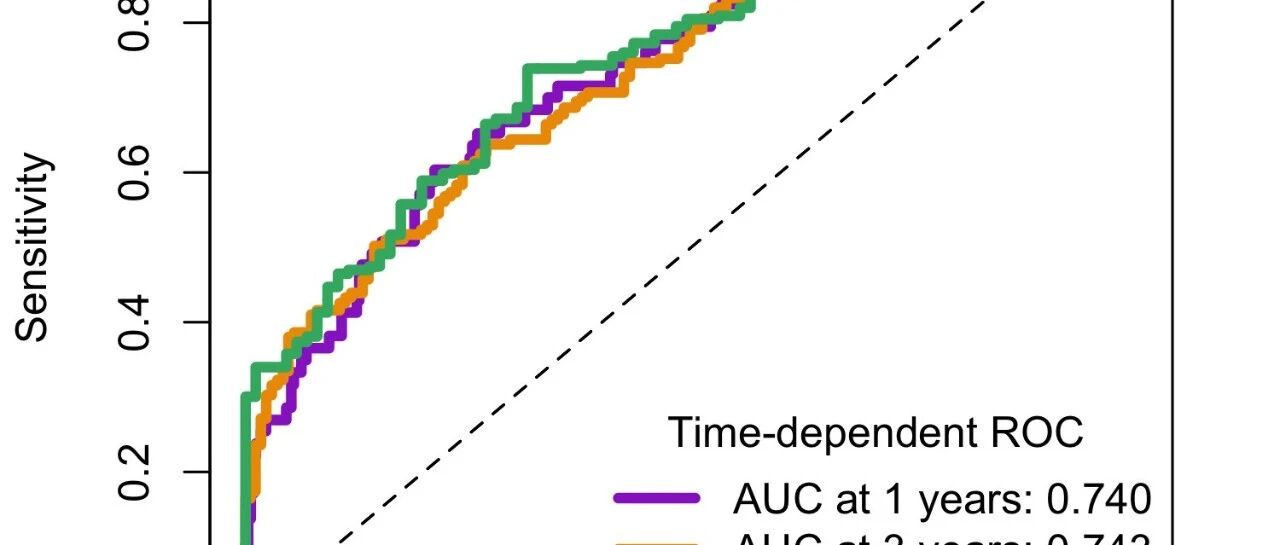
「图形解读」 :
-
「紫色曲线」 : 1年生存的ROC (AUC=0.740) -
「橙色曲线」 : 3年生存的ROC (AUC=0.743) -
「绿色曲线」 : 5年生存的ROC (AUC=0.764)
「关键发现」 :
-
「AUC随时间增加」 : 0.740 → 0.743 → 0.764 -
「5年预测最准确」 : AUC=0.764,判别能力较好 -
「模型稳定性好」 : 不同时间点AUC都>0.7
4.4 多变量ROC比较
比较riskScore与临床变量的预测性能:
# 定义预测时间点
predictTime <- 5# 5年
aucText <- c()
png(file = "picture/cliROC.all.png",
width = 5.5 * 300,
height = 5.5 * 300,
res = 300)
# 绘制riskScore的ROC
i <- 3
ROC_rt <- timeROC(
T = risk$time,
delta = risk$state,
marker = risk$riskScore,
cause = 1,
weighting = 'aalen',
times = c(predictTime),
ROC = TRUE
)
plot(ROC_rt,
time = predictTime,
col = bioCol[i - 2],
title = FALSE,
lwd = 4)
aucText <- c(paste0("Risk", ", AUC=", sprintf("%.3f", ROC_rt$AUC[2])))
# 添加对角参考线
abline(0, 1, lty = 2, col = "gray")
# 循环计算各临床变量的ROC
for (i in4:ncol(rt)) {
# 将因子变量转换为数值
marker_data <- as.numeric(as.factor(rt[, i]))
ROC_rt <- timeROC(
T = rt$time,
delta = rt$state,
marker = marker_data,
cause = 1,
weighting = 'aalen',
times = c(predictTime),
ROC = TRUE
)
plot(ROC_rt,
time = predictTime,
col = bioCol[i - 2],
title = FALSE,
lwd = 4,
add = TRUE)
aucText <- c(aucText, paste0(colnames(rt)[i], ", AUC=",
sprintf("%.3f", ROC_rt$AUC[2])))
}
# 添加图例
legend("bottomright",
aucText,
lwd = 4,
bty = "n",
col = bioCol[1:length(aucText)],
title = "5-year ROC",
cex = 0.8)
dev.off()
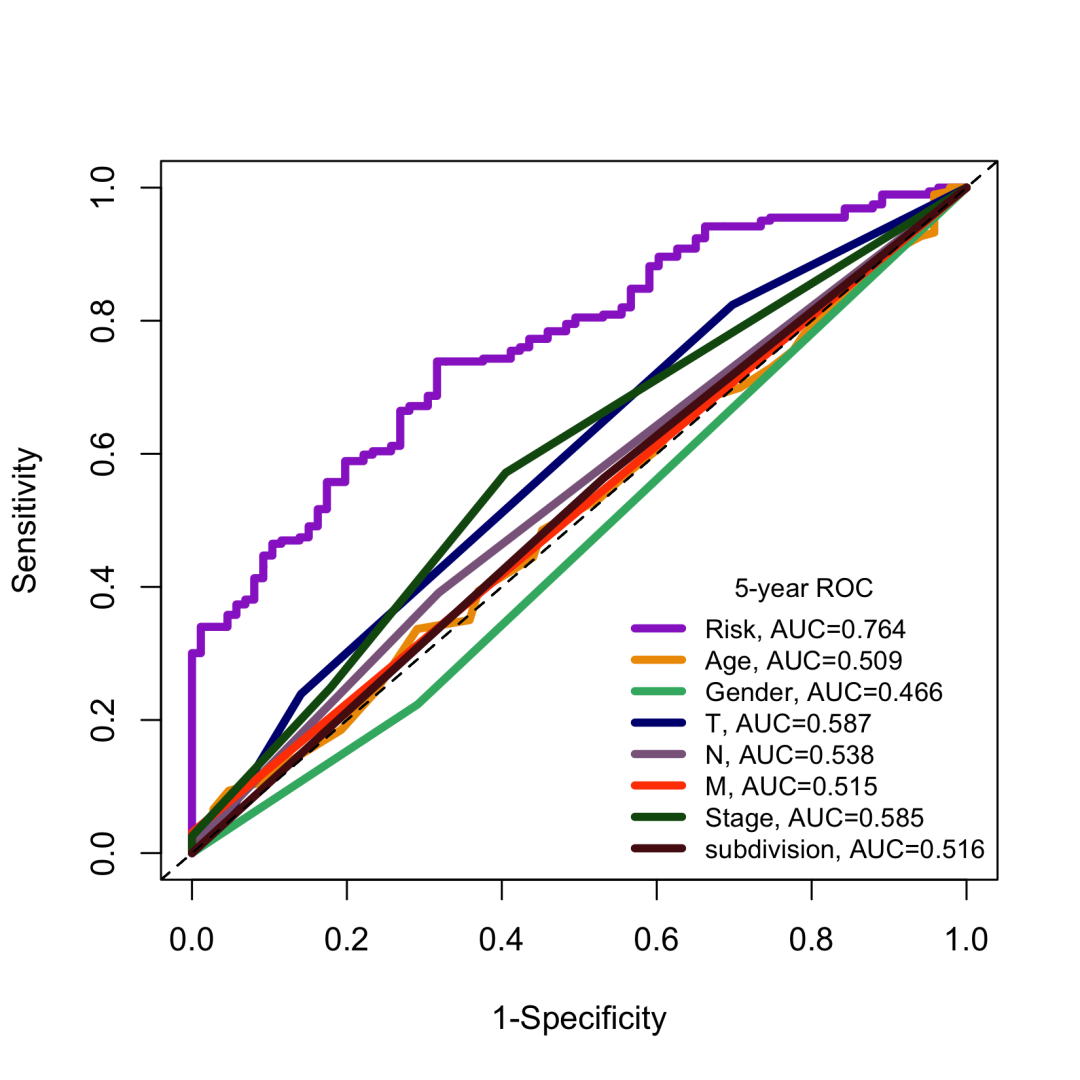
「图形解读」 :
各变量的5年AUC排名:
-
「Risk」 : 0.764 (最优) - 紫色 -
「T」 (肿瘤大小): 0.587 - 深蓝色 -
「Stage」 (临床分期): 0.585 - 深绿色 -
「N」 (淋巴结): 0.538 - 粉色 -
「subdivision」 : 0.516 - 棕色 -
「M」 (远处转移): 0.515 - 橙色 -
「Age」 : 0.509 - 黄色 -
「Gender」 : 0.466 - 浅绿色
「关键发现」 :
-
「Risk评分表现最佳」 (AUC=0.764),远超单个临床变量 -
「T和Stage」 是最好的临床预后因素(AUC≈0.59) -
「Gender」 预测能力最差(AUC=0.466,接近随机) -
「整合多个变量」 的Risk模型优于任何单一临床指标
4.5 计算最佳截断值
使用Youden指数确定最佳截断值:
# 计算最佳截断值
best_coords <- coords(roc1, "best", ret = c("threshold", "specificity", "sensitivity"))
cat("最佳截断值:", best_coords$threshold, "\n")
cat("对应灵敏度:", best_coords$sensitivity, "\n")
cat("对应特异度:", best_coords$specificity, "\n")
cat("Youden指数:", best_coords$sensitivity + best_coords$specificity - 1, "\n")
「结果」 :
-
最佳截断值: 0.840 -
灵敏度: 0.662 (66.2%) -
特异度: 0.662 (66.2%) -
Youden指数: 0.323
「应用」 :
-
riskScore > 0.840 → 高风险组 -
riskScore ≤ 0.840 → 低风险组 -
该截断值使灵敏度和特异度达到最佳平衡
5. 结果解读
5.1 模型性能评估
从我们的ROC分析中得到:
|
|
|
|
|---|---|---|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
「总体评价」 :
-
模型具有 「较好的判别能力」 (AUC=0.7-0.8) -
「长期预测优于短期」 (5年AUC最高) -
「优于所有单一临床变量」
5.2 与临床变量对比
Risk评分 vs 临床变量(5年AUC):
Risk: 0.764 ████████████████ T: 0.587 ████████████ Stage: 0.585 ████████████ N: 0.538 ███████████ Age: 0.509 ██████████ Gender:0.466 █████████ 「提升幅度」 :
-
vs T分期: 提升30% ((0.764-0.587)/0.587) -
vs Stage: 提升31% -
vs Gender: 提升64%
5.3 时间趋势分析
AUC随时间变化:
-
1年: 0.740 -
3年: 0.743 (+0.003) -
5年: 0.764 (+0.021)
「趋势解读」 :
-
AUC呈上升趋势 -
模型对 「长期预后预测更准确」 -
说明筛选的基因与长期生存密切相关
6. ROC曲线的优势与局限
6.2 局限性
-
「不考虑校准度」 :
-
只评估排序能力 -
需结合校准曲线评估 -
「类别不平衡」 :
-
正负样本比例极不平衡时可能失真 -
可考虑使用PR曲线 -
「缺乏临床意义」 :
-
AUC不直接反映临床获益 -
需结合DCA评估净获益 -
「生存数据特殊性」 :
-
删失数据处理复杂 -
需使用时间依赖ROC
7. 进阶分析
7.1 DeLong检验
比较两个ROC曲线的AUC是否有显著差异:
library(pROC)
# 构建另一个预测模型(如仅用Age)
roc2 <- roc(rt$state ~ rt$Age)
# DeLong检验
test_result <- roc.test(roc1, roc2, method = "delong")
cat("DeLong检验 p值:", test_result$p.value, "\n")
7.2 Bootstrap置信区间
估计AUC的稳健性:
# Bootstrap重采样(2000次)
ci_result <- ci.auc(roc1, method = "bootstrap", boot.n = 2000)
cat("AUC:", auc(roc1), "\n")
cat("95% CI:", ci_result[1], "-", ci_result[3], "\n")
7.3 决策曲线分析(DCA)
评估模型的临床净获益:
library(rmda)
# 构建决策曲线
dca_result <- decision_curve(
rt$state ~ rt$riskScore,
data = rt,
thresholds = seq(0, 1, 0.01),
confidence.intervals = 0.95
)
# 绘制DCA曲线
plot_decision_curve(dca_result,
curve.names = "Risk Score",
col = "red",
lwd = 2)
7.4 部分AUC(pAUC)
关注特定灵敏度或特异度范围:
# 计算特异度在80%-100%范围内的部分AUC
pauc <- auc(roc1, partial.auc = c(1, 0.8), partial.auc.focus = "sp")
cat("pAUC (sp 0.8-1.0):", pauc, "\n")
8. 注意事项
8.1 数据准备
「删失数据处理」 :
-
基本ROC: 将删失样本视为"阴性" -
时间依赖ROC: 正确处理删失(推荐)
「样本量要求」 :
-
至少需要100个样本 -
每组至少10个事件 -
样本越多,AUC估计越稳定
8.2 模型选择
「weighting参数」 (timeROC):
-
"aalen": Aalen加性模型(推荐) -
"marginal": Kaplan-Meier方法 -
"cox": Cox比例风险模型
「选择建议」 :
-
一般情况使用 "aalen" -
小样本使用 "marginal" -
比例风险假设成立用 "cox"
8.3 结果报告
「标准报告格式」 :
Risk Score showed good discrimination ability with AUC of 0.706 (95% CI: 0.659-0.754) for overall survival status. Time-dependent ROC analysis demonstrated AUC of 0.740, 0.743, and 0.764 for 1-year, 3-year, and 5-year OS, respectively.
送你一份科研小礼物,开启科研生涯!
点击即可领取: