大家好,今天继续讲KEGG富集分析
通过 KEGG通路富集分析 ,我们将系统性地探索与SERPINE1相关的差异表达基因参与了哪些重要的生物学通路,并用专业的可视化方法展示这些发现。
KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库是全球最权威的生物通路数据库之一,通过对差异表达基因进行KEGG富集分析,我们能够从分子水平理解SERPINE1在肿瘤发生发展中的功能机制,为后续的实验设计和临床应用提供重要指导。
1. 基因ID转换:构建分析基础
我们首先将基因符号转换为Entrez ID,这是进行KEGG富集分析的必要步骤。
# === 代码块 1:基因ID映射与转换 ===
library("clusterProfiler")
library("org.Hs.eg.db")
library("enrichplot")
library("ggplot2")
# 读取差异表达基因数据
gene_group_diff_matrix <- read.table("data/gene_grouping_diff_matrix.tsv",
header = TRUE, sep = "\t", check.names = FALSE)
# 提取基因名称并转换为Entrez ID
genes <- unique(as.vector(gene_group_diff_matrix[, 1]))
entrez_ids <- mget(genes, org.Hs.egSYMBOL2EG, ifnotfound = NA)
entrez_ids <- as.character(entrez_ids)
gene <- entrez_ids[entrez_ids != "NA"]
📈 运行结果
基因ID转换成功完成!我们从 382个输入基因 中成功获得了 318个有效的Entrez ID ,转换率达到83.2%。这为后续的通路富集分析提供了高质量的基因集合。
2. KEGG通路富集分析:系统性功能解读
利用clusterProfiler包进行全面的KEGG通路富集分析,识别显著富集的生物学通路。
# === 代码块 2:KEGG富集分析 ===
# 设置分析参数
pvalue_filter <- 0.05
qvalue_filter <- 0.05
# 执行KEGG富集分析
kk <- enrichKEGG(gene = gene, organism = "hsa", pvalueCutoff = 1, qvalueCutoff = 1)
KEGG <- as.data.frame(kk)
# 根据显著性阈值筛选结果
KEGG_filtered <- KEGG[(KEGG$pvalue < pvalue_filter & KEGG$qvalue < qvalue_filter),]
# 生成分析摘要
kegg_summary <- data.frame(
Total_Pathways = nrow(KEGG),
Significant_Pathways = nrow(KEGG_filtered),
P_Value_Threshold = pvalue_filter,
Q_Value_Threshold = qvalue_filter,
Total_Genes_Input = length(genes),
Valid_Entrez_IDs = length(gene)
)
运行结果
|
|
|
|---|---|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
结果解读:
富集分析结果令人振奋!在204个检测到的KEGG通路中,我们发现了 32个显著富集的通路 (P < 0.05, Q < 0.05),富集率高达15.7%。这表明与SERPINE1相关的差异表达基因具有明确的功能指向性。
3. 核心通路识别:解析关键生物学过程
深入分析排名前10的显著富集通路,揭示SERPINE1的核心功能机制。
# === 代码块 3:核心通路分析 ===
# 提取排名前10的显著通路
top_pathways <- head(KEGG_filtered[order(KEGG_filtered$pvalue), ], 10)
pathway_summary <- data.frame(
Pathway_ID = top_pathways$ID,
Pathway_Name = top_pathways$Description,
Gene_Count = top_pathways$Count,
P_Value = top_pathways$pvalue,
Q_Value = top_pathways$qvalue,
Gene_Ratio = top_pathways$GeneRatio
)
📈 运行结果
|
|
|
|
|
|
|---|---|---|---|---|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
结果解读:
这些核心通路揭示了SERPINE1的多重生物学功能:
-
炎症反应调控 :多个通路(AGE-RAGE、IL-17、细胞因子受体相互作用)均与炎症反应密切相关 -
细胞外基质重塑 :ECM-受体相互作用和蛋白质消化吸收通路显示其在组织重塑中的作用 -
免疫应答参与 :疟疾、阿米巴病等感染性疾病通路提示其在免疫反应中的重要性
🎨 4. 可视化展示:直观呈现富集结果
通过柱状图和点图两种方式展示KEGG富集分析结果。
# === 代码块 4:可视化绘制 ===
# 绘制富集通路柱状图
barplot(kk_filtered, drop = TRUE, showCategory = 20,
label_format = 30, color = "qvalue")
# 绘制富集通路点图
dotplot(kk_filtered, showCategory = 20, orderBy = "GeneRatio",
label_format = 30, color = "qvalue")
📊 运行结果
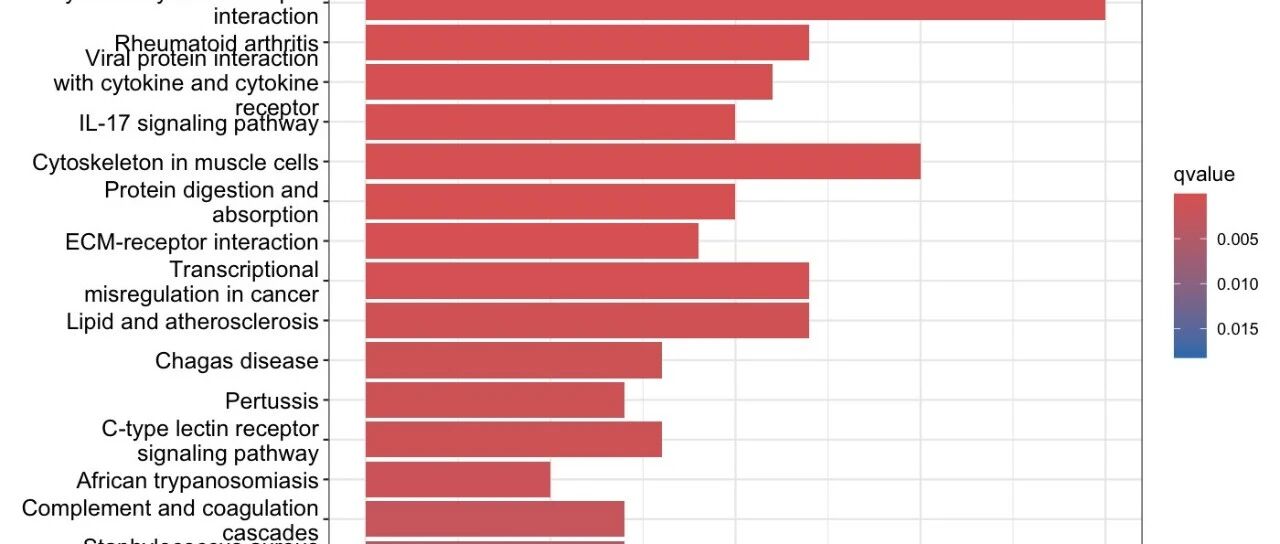
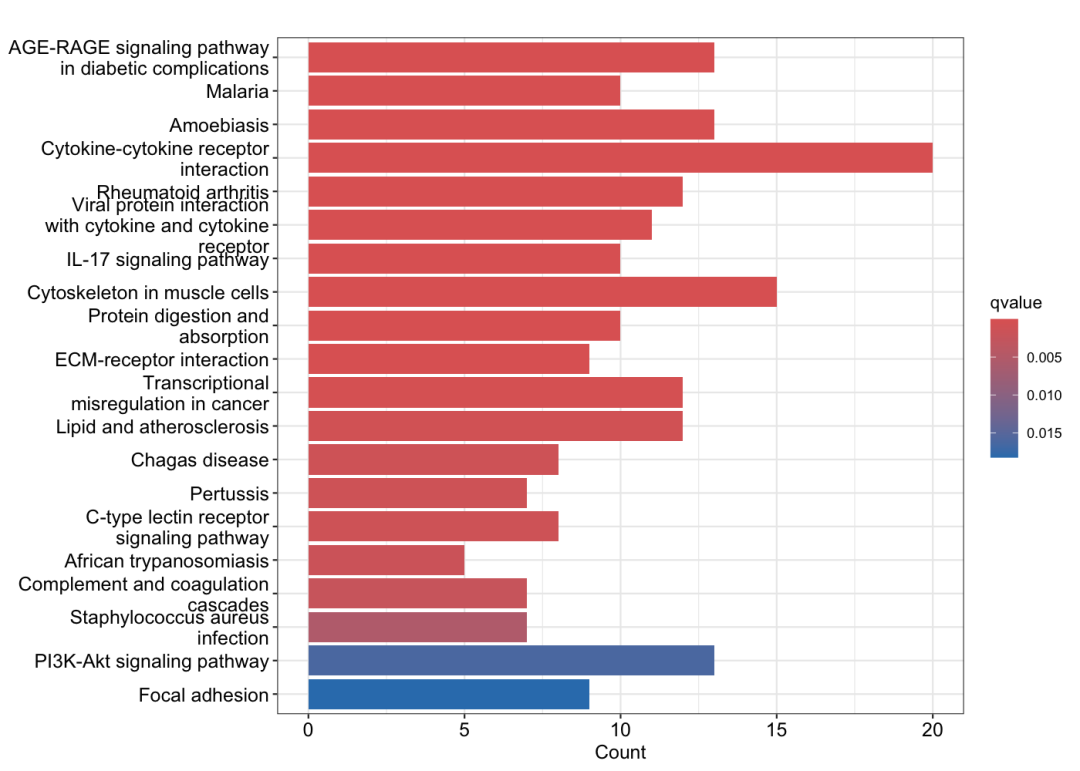
结果解读:
柱状图清晰地展示了各个显著富集通路的统计学意义。柱子的长度代表富集的基因数量,颜色深浅反映Q值的大小(颜色越深,显著性越高)。我们可以看到:
-
AGE-RAGE信号通路 和 细胞因子-细胞因子受体相互作用 通路的富集基因数最多 -
大部分显著通路的Q值都极小(< 1e-05),显示出极强的统计学显著性 -
通路功能涵盖了从炎症反应到组织重塑的多个生物学过程
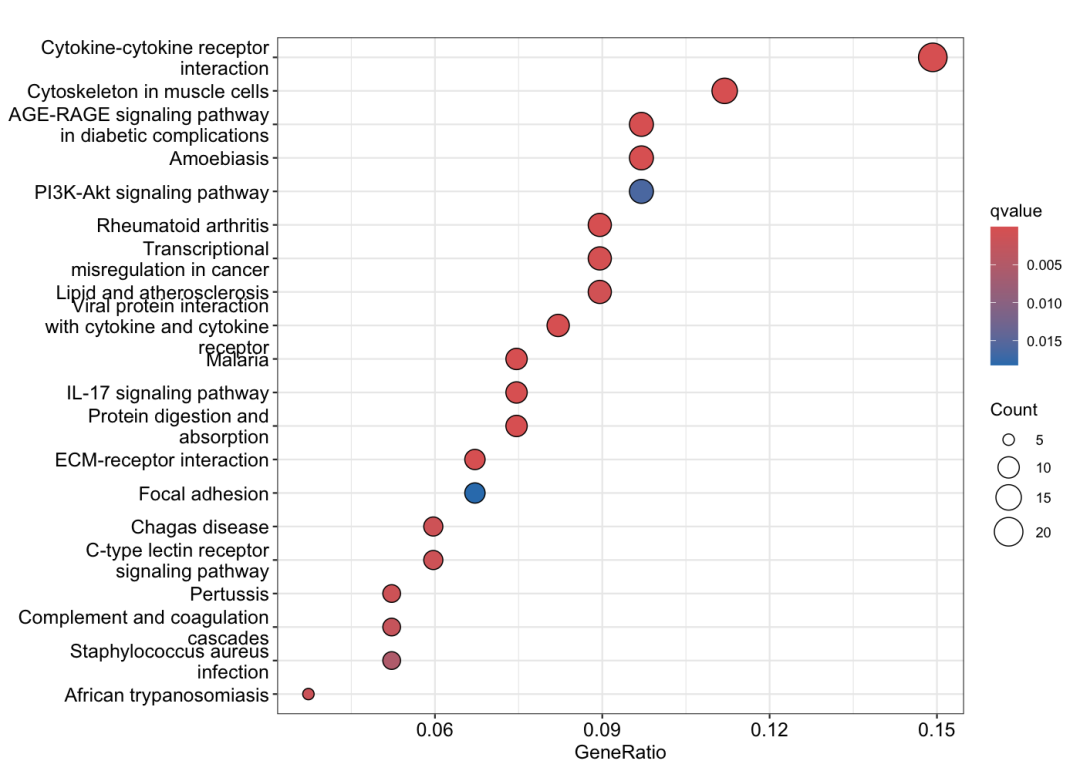
结果解读:
点图提供了更丰富的信息维度:
-
X轴(GeneRatio) :表示该通路中富集基因占输入基因的比例 -
点的大小 :代表富集的基因数量 -
颜色深浅 :反映Q值的显著性水平
从图中可以观察到,虽然某些通路(如疟疾、阿米巴病)的基因比例较高,但 细胞因子-细胞因子受体相互作用 通路由于富集基因数量最多,在功能网络中可能占据更重要的地位。
5. 功能机制解读:从通路到生物学意义
基于富集分析结果,我们可以构建SERPINE1的功能调控网络模型。
核心功能模块:
-
炎症调控模块 :AGE-RAGE信号通路、IL-17信号通路等,提示SERPINE1在慢性炎症和组织损伤中发挥重要作用
-
免疫应答模块 :病原体感染相关通路(疟疾、阿米巴病)显示其在宿主免疫防御中的参与
-
组织重塑模块 :ECM-受体相互作用、胶原蛋白代谢等通路揭示其在组织纤维化和创伤愈合中的功能
-
细胞通信模块 :细胞因子网络相关通路表明其作为细胞间信号传导的重要媒介
完整代码联系老师免费获取(备注“TCGA KEGG”)

以上就是今天的内容,希望对你有帮助!欢迎点赞、在看、关注、转发。