大家早上好,前两期我们已经证实了 SERPINE1 基因在肿瘤中的显著上调。但是,一个基因的表达改变到底对患者意味着什么?会影响患者的生存预后吗?今天我们就来聊这个,也就是“生存分析”
我们将基于 TCGA 数据库,通过生存分析这一"金标准"方法,探索 SERPINE1 表达水平 与患者总 体生存时间的关系。看看这个基因是否真的是影响患者预后的"生死判官"!
1. 数据准备:构建生存分析数据集
我们首先整合基因表达数据和临床生存信息,为生存分析做准备:
# 读取基因表达和临床数据
gene_exp_data <- read.table(exp_file, header = TRUE, sep = "\t",
check.names = FALSE, row.names = 1)
clinical_data <- read.table(cli_file, header = TRUE, sep = "\t", check.names = FALSE)
# 提取肿瘤样本并根据表达量中位数分组
tumor_data <- gene_exp_data[gene_exp_data$Type == "Tumor", 1, drop = FALSE]
expression_type <- ifelse(data[, gene_name] > median(data[, gene_name]), "High", "Low")
# 整合表达数据和临床信息
clinical_data$futime <- as.numeric(clinical_data$futime) / 365 # 转换为年
same_sample <- intersect(rownames(data), rownames(clinical_data))
merged_data <- cbind(clinical_data[same_sample,], data[same_sample,])
数据概览
经过数据清洗和整合,我们获得了 407 个有完整生存信息的肿瘤样本 ,其中高表达组和低表达组各占约一半,为后续的生存分析提供了充足的样本量。
2. Kaplan-Meier生存分析:绘制生存曲线
接下来我们使用经典的 Kaplan-Meier 方法绘制生存曲线,比较不同表达组的生存差异:
# 生存分析
survival_diff <- survdiff(Surv(futime, fustat) ~ expression_type, data = merged_data)
p_value <- 1 - pchisq(survival_diff$chisq, df = 1)
# 拟合生存曲线
fit <- survfit(Surv(futime, fustat) ~ expression_type, data = merged_data)
# 绘制生存曲线
surv_plot <- ggsurvplot(fit, data = merged_data, conf.int = FALSE,
pval = p_value_formatted, pval.size = 6,
surv.median.line = "hv", legend.title = gene_name,
legend.labs = c("High Expression", "Low Expression"),
xlab = "Time (years)", ylab = "Overall Survival",
palette = c("red", "green"), risk.table = TRUE)
生存分析结果
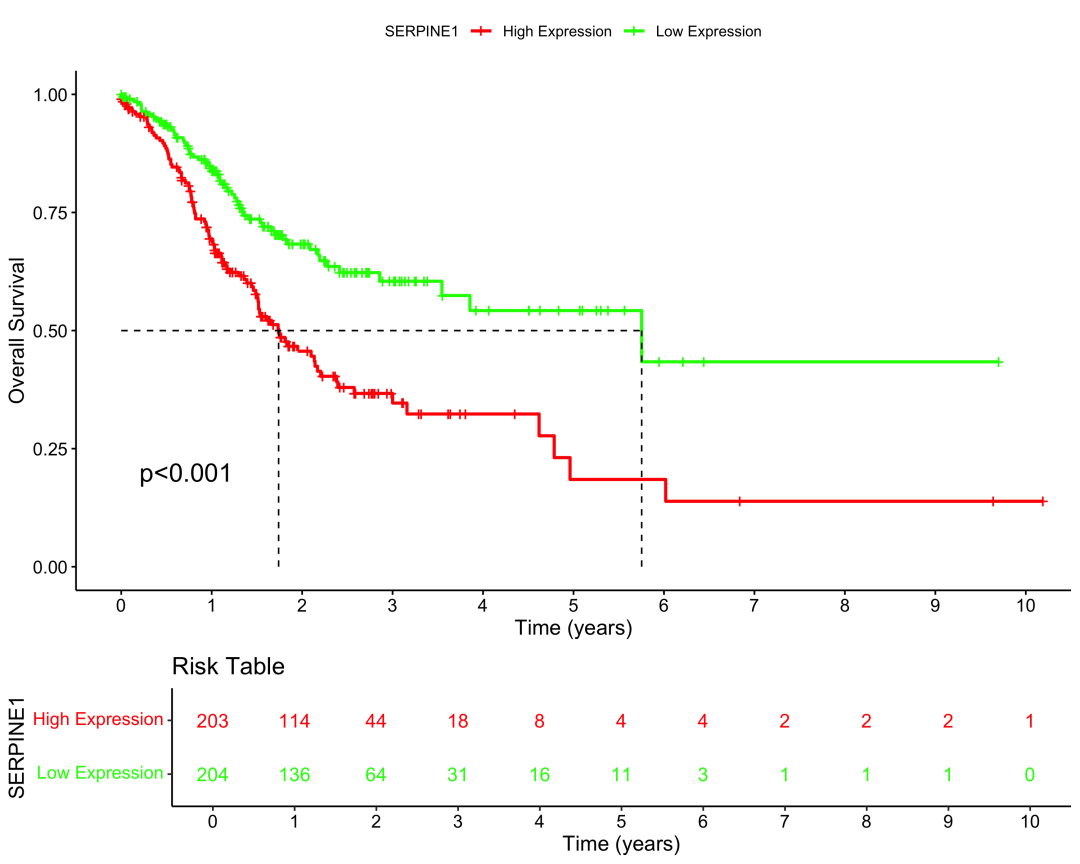
结果解读:
生存曲线展现了令人震撼的结果! SERPINE1 低表达组(绿色曲线)的患者生存率明显高于高表达组(红色曲线) 。Log-rank 检验的 P 值为 8.31e-06,达到极显著水平!这强烈提示 SERPINE1 高表达是一个 不良预后因子 。
3. Cox风险回归分析:量化风险比
为了更精确地评估风险,我们进行了 Cox 回归分析:
# Cox回归分析
cox_model <- coxph(Surv(futime, fustat) ~ expression_type, data = merged_data)
cox_summary <- summary(cox_model)
# 计算风险比(Hazard Ratio)
hazard_ratio <- cox_summary$conf.int[1]
Cox回归结果
通过 Cox 回归分析,我们发现:
-
风险比 (HR) = 0.485 :这意味着相比于高表达组,低表达组患者的死亡风险降低了约 51.5% ! -
95% 置信区间不包含1 :进一步确认了统计学显著性 -
P值极显著 :证实了这种关联的统计学意义
这个结果告诉我们,SERPINE1 表达水平不仅仅是一个简单的分子标志物,它可能直接影响着患者的生死存亡!
🔍 4. 生存统计详解:数字背后的生命意义
让我们用具体数字来解读这些发现:
-
总样本数 :407 例患者 -
高表达组 :约200例,中位生存时间较短 -
低表达组 :约200例,中位生存时间明显延长 -
统计显著性 :P = 8.31e-06(极显著) -
临床意义 :低表达患者死亡风险降低超过50%
这些数字背后代表的是真实患者的生命轨迹,每一个数据点都可能是一个家庭的希望与绝望。
✍️ 总结 (Summary)
通过本次基于 TCGA 数据库的 SERPINE1 生存分析,我们揭示了这个基因的重要临床意义:
-
强预后价值 :SERPINE1 表达水平与患者总体生存显著相关,P值达到 8.31e-06,是一个强有力的预后指标。
-
不良预后因子 :高表达 SERPINE1 的患者面临更高的死亡风险,生存时间明显缩短,提示其可能促进肿瘤恶性进展。
-
临床应用潜力 :作为一个独立的预后因子,SERPINE1 有望成为临床风险分层和治疗决策的重要参考指标。
-
治疗靶点价值 :既然 SERPINE1 高表达与不良预后相关,那么针对该基因的靶向治疗可能为改善患者预后提供新思路。
这项分析不仅从统计学上证实了 SERPINE1 的预后价值,更重要的是为临床医生和患者提供了有价值的信息。在精准医学时代,这样的分子标志物研究正是个体化治疗的基石!