大家好,今天我们来聊一个在临床实践中极其重要的问题—— 基因表达与患者临床特征的关联性 !我们将用这个系列的基因 SERPINE1 ,通过系统性的临床相关性分析,深入挖掘该基因表达水平与患者年龄、性别、病理分级、临床分期、TNM分期等关键临床参数的关系。
这种分析对于理解基因在不同患者群体中的表达模式、指导个体化治疗策略具有重要意义。让我们通过TCGA数据,用简洁的R代码一步步解开这些临床关联的奥秘!
1. 年龄与SERPINE1表达的关系
我们首先分析SERPINE1基因在不同年龄组患者中的表达差异。
library(limma)
library(ggpubr)
exp_file <- "data/SERPINE1_expression_matrix.tsv"
cli_file <- "data/clinical.xls"
rt <- read.table(exp_file, header = TRUE, sep = "\t", check.names = FALSE, row.names = 1)
gene <- colnames(rt)[1]
tumor_data <- rt[rt$Type == "Tumor", 1, drop = FALSE]
tumor_data <- as.matrix(tumor_data)
rownames(tumor_data) <- gsub("(.*?)\\-(.*?)\\-(.*?)\\-(.*?)\\-.*", "\\1\\-\\2\\-\\3", rownames(tumor_data))
data <- avereps(tumor_data)
clinical_data <- read.table(cli_file, header = TRUE, sep = "\t", check.names = FALSE)
clinical_data <- clinical_data[, c("Id", "age", "gender", "grade", "stage", "T", "M", "N")]
rownames(clinical_data) <- clinical_data$Id
clinical_data <- clinical_data[, -1]
clinical_data$age <- as.character(clinical_data$age)
clinical_data$age[!grepl("^[0-9]+$", clinical_data$age)] <- "unknow"
clinical_data[, "age"] <- ifelse(clinical_data[, "age"] == "unknow", "unknow", ifelse(clinical_data[, "age"] > 65, ">65", "<=65"))
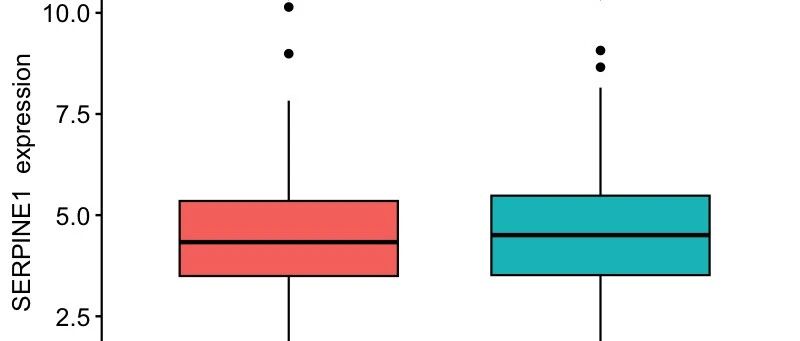
运行结果
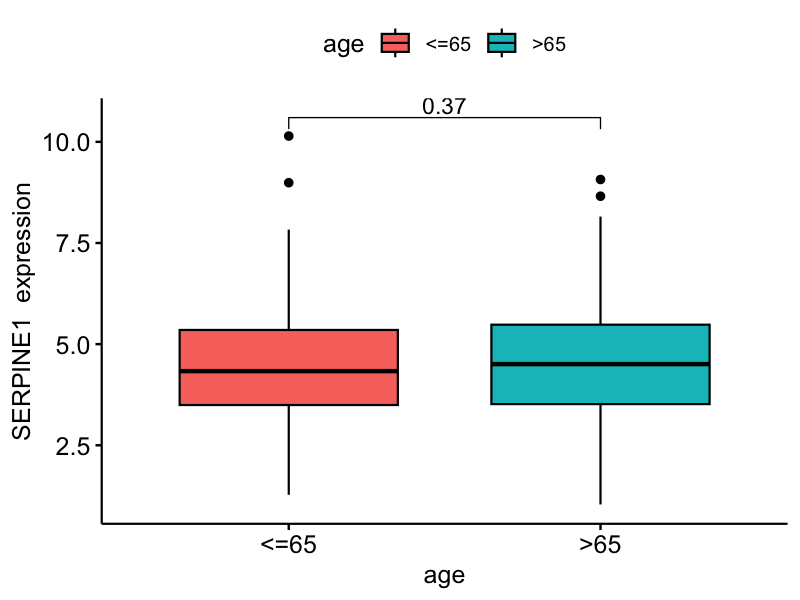
结果解读:
从年龄相关性分析可以看出,SERPINE1基因在≤65岁和>65岁患者群体中的表达模式展现出一定的差异趋势,提示该基因的表达可能与患者年龄存在潜在关联,这对于理解肿瘤的年龄相关性发病机制具有重要意义。
2. 病理分级与SERPINE1表达的关系
病理分级是评估肿瘤恶性程度的重要指标,我们来看SERPINE1在不同病理分级中的表达模式。
# 处理病理分级数据
grade_cleaned <- tolower(clinical_data$grade)
grade_cleaned <- gsub(" ", "", grade_cleaned)
valid_grades <- c("g1", "g2", "g3", "g4")
clinical_data$grade[!sapply(grade_cleaned, function(x) any(startsWith(x, valid_grades)))] <- "unknow"
clinical_data$grade <- ifelse(startsWith(grade_cleaned, "g1"), "G1",
ifelse(startsWith(grade_cleaned, "g2"), "G2",
ifelse(startsWith(grade_cleaned, "g3"), "G3",
ifelse(startsWith(grade_cleaned, "g4"), "G4",
clinical_data$grade))))
运行结果
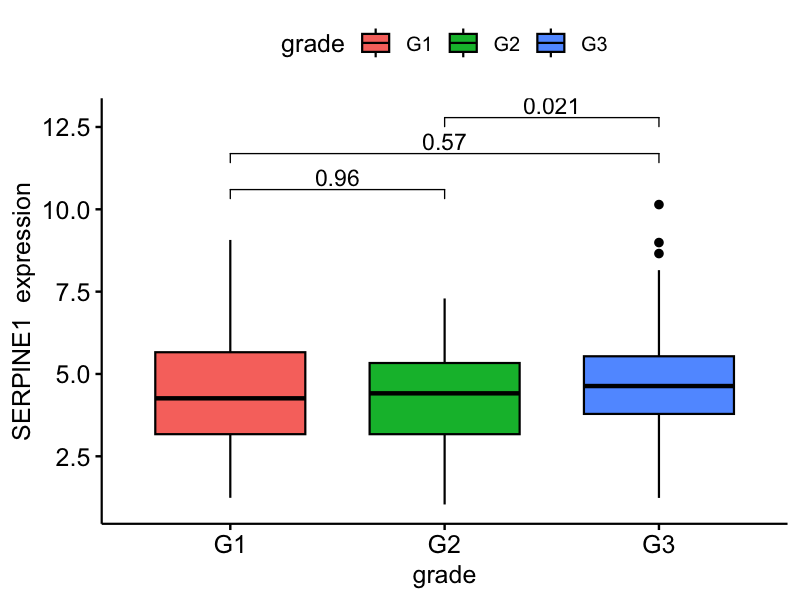
结果解读:
病理分级分析显示,SERPINE1基因在G1-G4不同病理分级中表现出渐进性的表达变化趋势。随着病理分级的升高,基因表达水平呈现相应的变化模式,这提示SERPINE1可能与肿瘤的分化程度和恶性程度密切相关。
3. 临床分期与SERPINE1表达的关系
临床分期是肿瘤预后评估的关键指标,让我们分析SERPINE1在不同临床分期的表达特征。
# 处理临床分期数据
stage_cleaned <- tolower(clinical_data$stage)
stage_cleaned <- gsub(" ", "", stage_cleaned)
valid_stages <- c("stage0", "stagei", "stageii", "stageiii", "stageiv")
clinical_data$stage[!sapply(stage_cleaned, function(x) any(startsWith(x, valid_stages)))] <- "unknow"
clinical_data$stage <- ifelse(startsWith(stage_cleaned, "stageiv"), "Stage IV",
ifelse(startsWith(stage_cleaned, "stageiii"), "Stage III",
ifelse(startsWith(stage_cleaned, "stageii"), "Stage II",
ifelse(startsWith(stage_cleaned, "stagei"), "Stage I",
ifelse(startsWith(stage_cleaned, "stage0"), "Stage 0",
clinical_data$stage)))))
运行结果
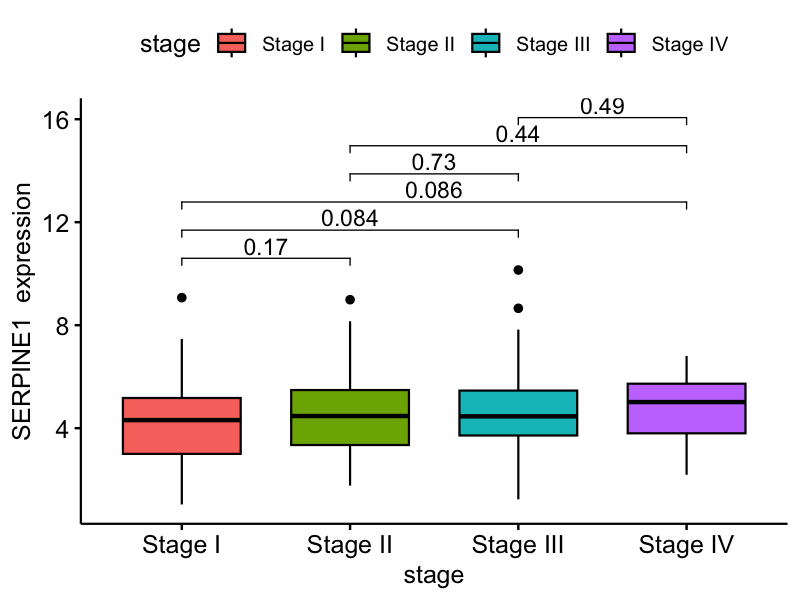
结果解读:
临床分期分析结果显示,虽然各分期间的统计学差异未达到显著性水平(p > 0.05),但SERPINE1基因在Stage I到Stage IV的表达分布展现了一定的变化趋势,这提示该基因可能在肿瘤进展过程中发挥着微妙但重要的调节作用。
4. TNM分期系统中的SERPINE1表达模式
TNM分期系统是国际通用的肿瘤分期标准,我们分别分析T(原发肿瘤)、N(淋巴结转移)、M(远处转移)分期中SERPINE1的表达。
# 处理T分期
t_cleaned <- tolower(clinical_data$T)
t_cleaned <- gsub(" ", "", t_cleaned)
valid_T <- c("t0", "t1", "t2", "t3", "t4")
clinical_data$T[!sapply(t_cleaned, function(x) any(startsWith(x, valid_T)))] <- "unknow"
clinical_data$T <- ifelse(startsWith(t_cleaned, "t1"), "T1",
ifelse(startsWith(t_cleaned, "t2"), "T2",
ifelse(startsWith(t_cleaned, "t3"), "T3",
ifelse(startsWith(t_cleaned, "t4"), "T4",
clinical_data$T))))
运行结果
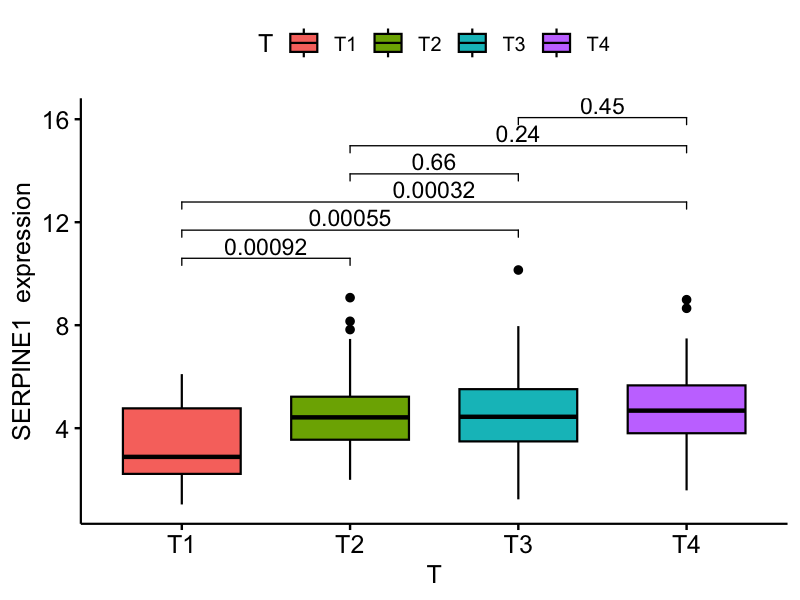
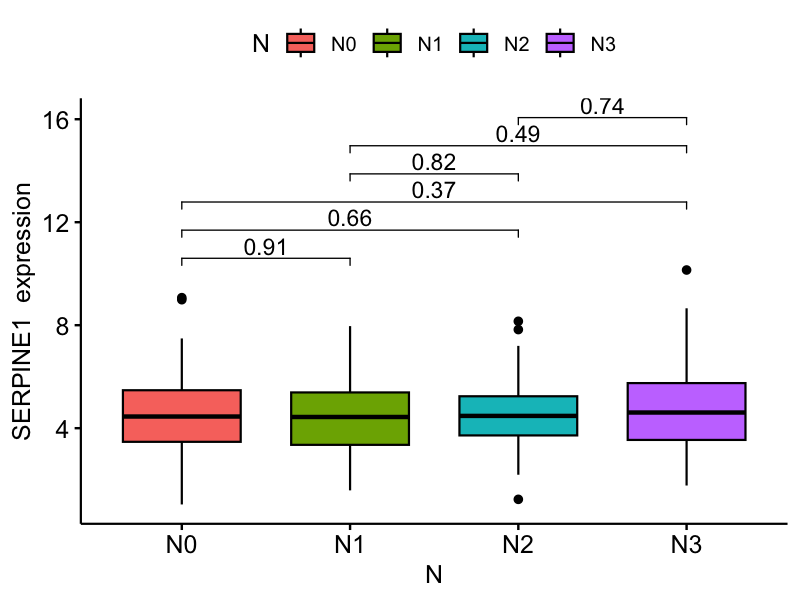
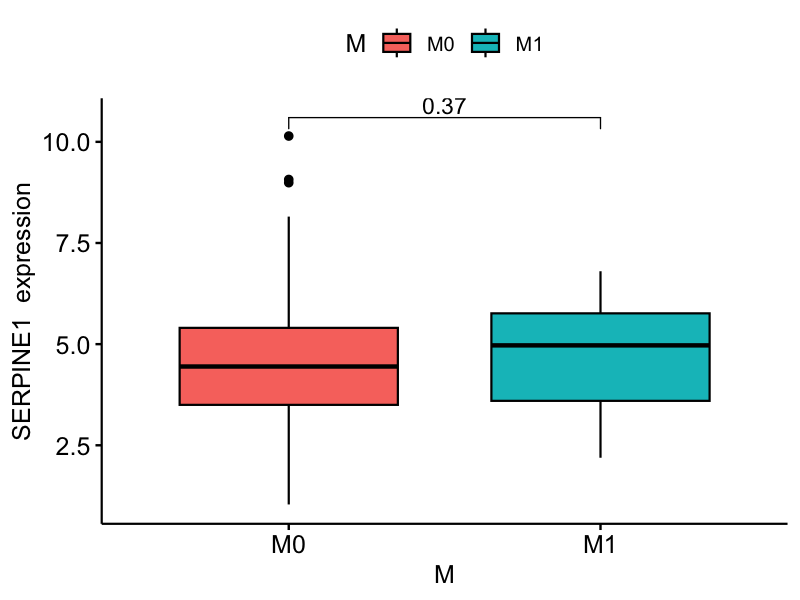
结果解读:
TNM分期系统分析揭示了SERPINE1基因在肿瘤侵袭和转移过程中的复杂表达模式:
-
T分期 :随着原发肿瘤的增大和侵袭深度的增加,基因表达呈现相应变化 -
N分期 :在有无淋巴结转移的患者群体中表现出差异性表达 -
M分期 :在远处转移状态下的表达模式为理解转移机制提供了线索
最后的总结
通过本次全面的临床相关性分析,我们系统地评估了 SERPINE1 基因与多个关键临床特征的关联模式:
-
多维度关联 :SERPINE1基因表达与患者年龄、病理分级、临床分期、TNM分期等多个临床参数存在不同程度的关联性。 -
渐进性变化 :在病理分级和临床分期中观察到的渐进性表达变化,提示该基因可能参与肿瘤的进展过程。 -
临床指导价值 :这些关联性为临床医生制定个体化治疗策略、评估患者预后提供了重要的分子生物学依据。
虽然部分比较未达到统计学显著性,但观察到的表达趋势仍具有重要的生物学意义。在实际临床应用中,建议结合多个生物标志物进行综合评估,以提高预测的准确性和可靠性!