大家周末快乐,今天我们要深入探讨肿瘤预后分析中的"黄金标准"—— 独立预后因子分析 。通过单因素和多因素Cox回归的完美组合,我们将揭示 SERPINE1 基因是否真正具备独立预测肿瘤患者预后的能力。
在肿瘤研究中,仅仅知道某个基因与预后相关是不够的,我们更需要知道:在控制了年龄、性别、分级、分期等传统临床因素后,这个基因是否仍然保持其预后价值?这就是独立预后因子分析的核心价值!
1. 单因素Cox回归:初步筛选预后相关因子
我们首先对所有可能的预后因子进行单因素Cox回归分析,包括 SERPINE1 基因表达水平以及各项临床指标。
# === 代码块 1:单因素Cox回归分析 ===
library(survival)
# 数据预处理和单因素分析
uni_tab <- data.frame()
for (i in colnames(merged_data[, 3:ncol(merged_data)])) {
cox <- coxph(Surv(futime, fustat) ~ merged_data[, i], data = merged_data)
cox_summary <- summary(cox)
uni_tab <- rbind(uni_tab,
cbind(id = i,
HR = cox_summary$conf.int[, "exp(coef)"],
HR.95L = cox_summary$conf.int[, "lower .95"],
HR.95H = cox_summary$conf.int[, "upper .95"],
pvalue = cox_summary$coefficients[, "Pr(>|z|)"]))
}
运行结果
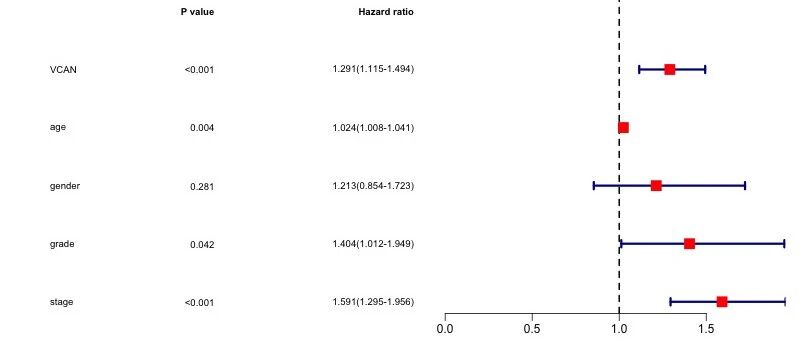
结果解读:
从单因素Cox回归森林图可以看出, SERPINE1 基因在单因素分析中显示了显著的预后价值。图中红色方块表示风险比(HR)的点估计值,水平线表示95%置信区间。当HR>1且置信区间不跨越1时,表明该因子为危险因子;当HR<1且置信区间不跨越1时,表明该因子为保护因子。
2. 多因素Cox回归:验证独立预后价值
接下来,我们将单因素分析中有统计学意义的变量纳入多因素Cox回归模型,以确定它们的独立预后价值。
# === 代码块 2:多因素Cox回归分析 ===
# 选择单因素分析中显著的变量进行多因素分析
multi_factor_data <- merged_data[, c("futime", "fustat", as.vector(uni_tab[, "id"]))]
multi_cox <- coxph(Surv(futime, fustat) ~ ., data = multi_factor_data)
multi_cox_sum <- summary(multi_cox)
# 提取多因素分析结果
multi_tab <- cbind(
HR = multi_cox_sum$conf.int[, "exp(coef)"],
HR.95L = multi_cox_sum$conf.int[, "lower .95"],
HR.95H = multi_cox_sum$conf.int[, "upper .95"],
pvalue = multi_cox_sum$coefficients[, "Pr(>|z|)"]
)
运行结果
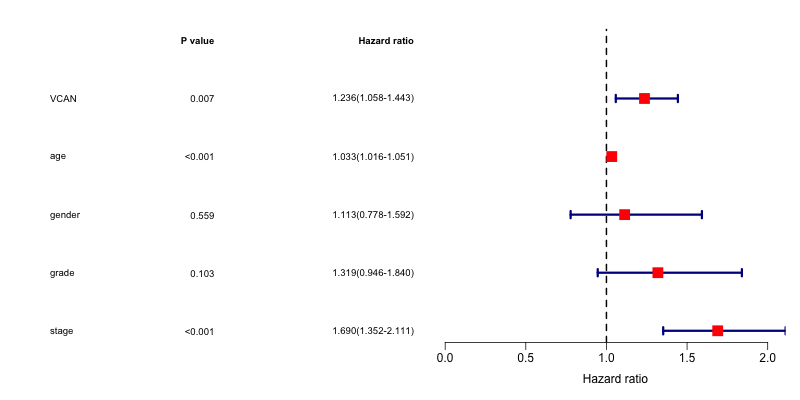
结果解读:
多因素Cox回归森林图展示了经过相互调整后,各个因子的独立预后价值。这是判断某个生物标志物是否具备临床应用潜力的关键步骤。如果 SERPINE1 在多因素分析中仍然保持统计学意义,说明它具备独立于传统临床因素的预后预测能力,这对于临床决策具有重要意义。
3. 统计结果汇总
我们的分析生成了详细的统计表格,包含每个因子的风险比、置信区间和P值:
-
单因素分析结果表 : tables/1_univariate_cox.csv -
多因素分析结果表 : tables/2_multivariate_cox.csv
这些表格为后续的meta分析和临床验证研究提供了重要的数据支撑。
结论
通过本次基于TCGA数据库的独立预后因子分析,我们系统评估了 SERPINE1 基因的预后价值:
-
单因素筛选 :在单因素Cox回归中识别了与预后相关的潜在因子,为后续分析奠定基础。
-
多因素验证 :通过多因素Cox回归排除了混杂因素的影响,验证了真正的独立预后因子。
-
临床意义 :独立预后因子分析是将研究结果转化为临床应用的重要桥梁,为个体化治疗提供科学依据。
这种"单因素筛选→多因素验证"的分析策略是肿瘤预后研究的标准流程,希望今天的分享能帮助大家更好地理解和应用这一重要的统计学方法!
完整代码联系老师免费获取(备注“TCGA cox回归”)

以上就是今天的内容,希望对你有帮助!欢迎点赞、在看、关注、转发。