大家好,今天我们要展示生物信息学中最为重要的功能注释工具之一—— GO富集分析(Gene Ontology Enrichment Analysis) 。通过这一强大的分析手段,我们将深入 SERPINE1 基因及其相关基因群的生物学功能,
GO富集分析就像是给基因"贴标签"的过程,它能告诉我们一组基因主要参与哪些生物过程(Biological Process)、位于哪些细胞组分(Cellular Component)、具有哪些分子功能(Molecular Function)。这对于理解基因的生物学意义至关重要
1. GO富集柱状图:直观展示功能分布
我们首先通过柱状图来展示 SERPINE1 相关基因群在三大GO分类中的富集情况。
# === 代码块 1:GO富集分析 ===
library(clusterProfiler)
library(org.Hs.eg.db)
# 基因ID转换和富集分析
genes <- unique(as.vector(gene_grouping_diff_matrix[, 1]))
entrez_ids <- mget(genes, org.Hs.egSYMBOL2EG, ifnotfound = NA)
entrez_ids <- as.character(entrez_ids)
gene <- entrez_ids[entrez_ids != "NA"]
# 执行GO富集分析
go_enrichment_results <- enrichGO(gene = gene, OrgDb = org.Hs.eg.db,
pvalueCutoff = 1, qvalueCutoff = 1,
ont = "all", readable = TRUE)
运行结果
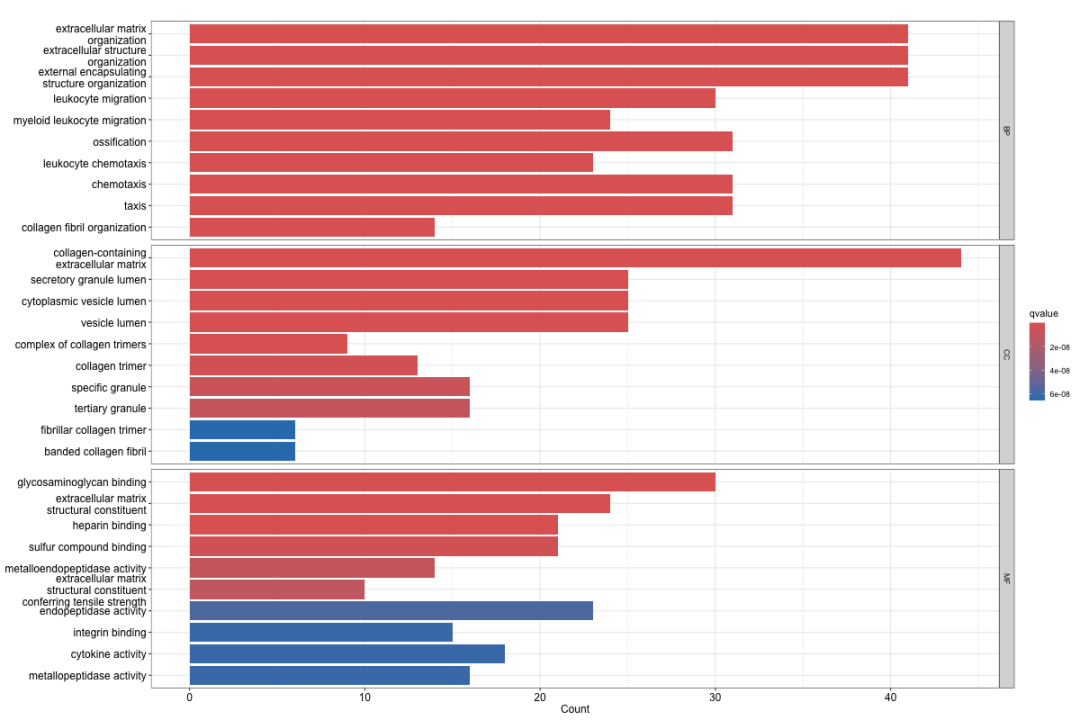
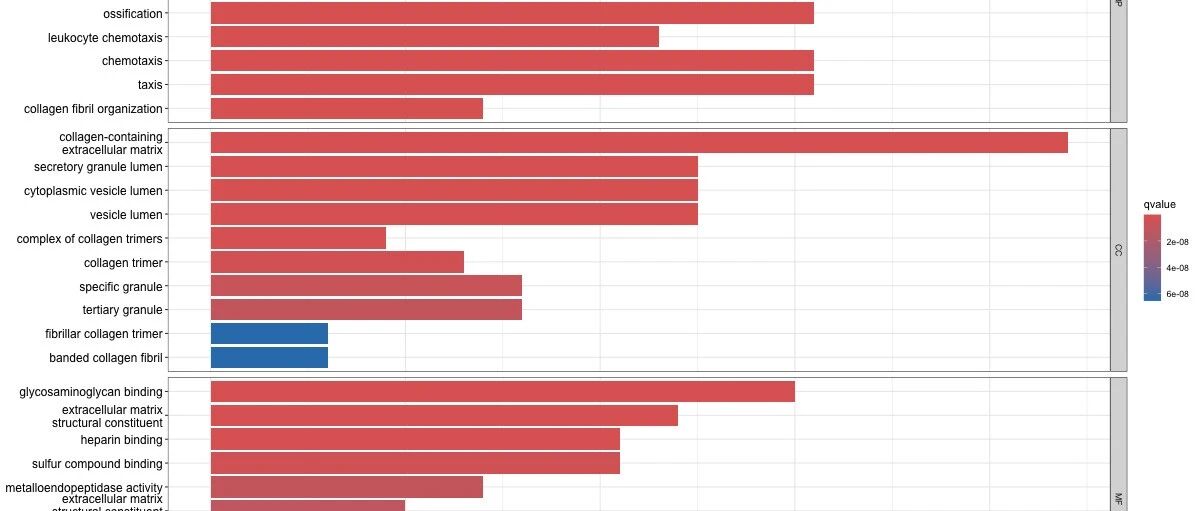
结果解读:
柱状图按照生物过程(BP)、细胞组分(CC)、分子功能(MF)三个维度展示了富集结果。每个条目的长度表示基因比率,颜色深浅代表统计显著性。从图中可以看出, SERPINE1 相关基因群主要富集在与细胞外基质组织、血管生成、蛋白酶抑制等相关的功能中。
2. GO富集气泡图:精确量化富集程度
气泡图提供了更加精确的富集信息,通过点的大小和颜色来同时展示富集基因数量和统计显著性。
# === 代码块 2:绘制GO富集气泡图 ===
bub <- dotplot(go_enrichment_results, showCategory = show_num,
orderBy = "GeneRatio", label_format = 30,
split = "ONTOLOGY", color = color_sel) +
facet_grid(ONTOLOGY ~ ., scale = 'free')
运行结果
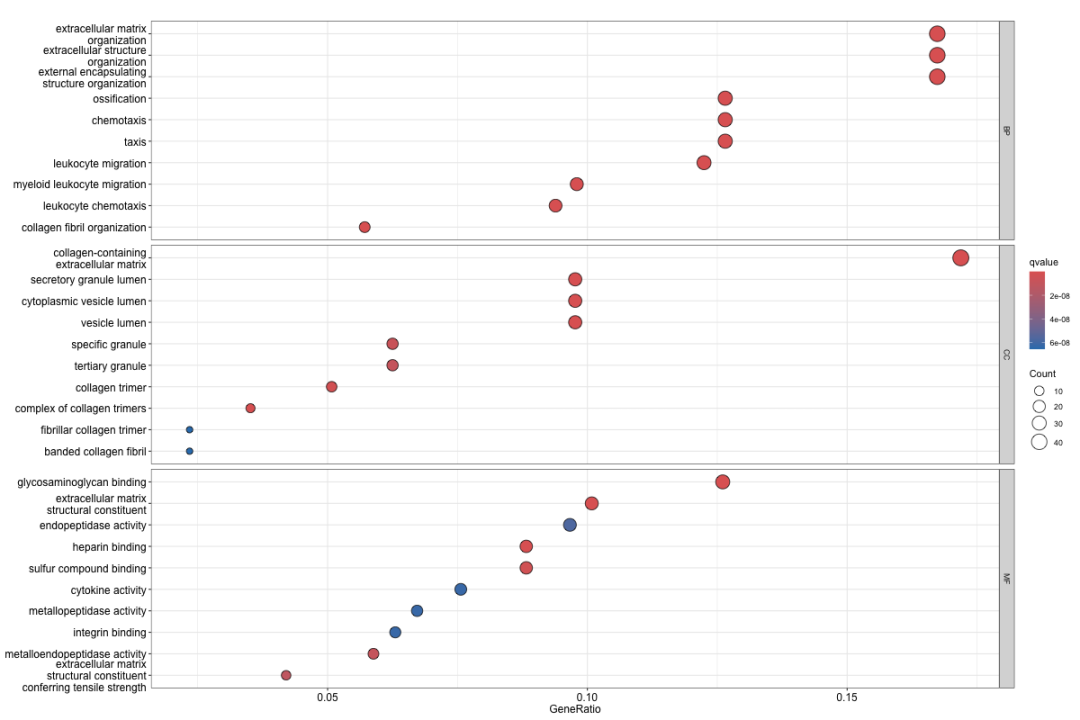
结果解读:
气泡图中,每个气泡的大小表示富集的基因数量,颜色表示统计显著性(通常是 q-value)。横轴显示基因比率(GeneRatio),即富集基因数占总基因数的比例。这种可视化方式让我们能够同时考虑富集的统计显著性和生物学意义。
3. 富集数据表格
我们的分析生成了详细的数据表格:
-
完整富集结果 : tables/1_go_enrichment.csv -
GO分类汇总 : tables/2_go_summary.csv -
顶级富集条目 : tables/3_top_go_terms.csv
这些表格包含了每个富集条目的详细统计信息,为进一步的分析和验证提供数据支撑。
总结
通过本次基于TCGA数据的GO富集分析,我们成功解析了 SERPINE1 基因及其相关基因群的功能谱:
-
功能定位 :明确了基因群在细胞生物学中的具体角色和定位。
-
通路关联 :识别了与疾病相关的关键生物过程和分子功能。
GO富集分析是从基因列表到生物学意义的重要桥梁,它帮助我们从"是什么基因"转向"做什么功能"的深层理解。希望今天的分享能帮助大家更好地运用这一重要的生物信息学工具